En este artículo científico, se resumen varios estudios de laboratorio sobre estrategias de adaptación evolutivas de bacterias bajo antibióticos, como la resistencia y la tolerancia.
Presentación de las estrategias evolutivas de bacterias bajo antibióticos
“Bacteria under antibiotic attack: Different strategies for evolutionary adptatition” (“Bacterias bajo el ataque de antibióticos: Diferentes estrategias de adaptación evolutiva”), se trata de un artículo de carácter científico que reúne un resumen de varios estudios científicos, elaborado por Etthel M. Windels, Bram Van den Bergh y Jan Michiels, y publicado en la revista científica Plos Pathogens en Mayo de 2020; el cual versa sobre las distintas estrategias de adaptación evolutivas observadas en estudios de bacterias bajo antibióticos, tales como la resistencia y la tolerancia.
Las bacterias son unos microorganismos bien conocidos por su extremadamente alta adaptabilidad a entornos estresantes. La relevancia clínica de esta propiedad, queda claramente ilustrada por la eficacia cada vez menor de las terapias con antibióticos. Las exposiciones frecuentes a los antibióticos, favorecen a las cepas bacterianas que han adquirido mecanismos para superar la inhibición y la letalidad de los fármacos. Muchas cepas, incluidos los patógenos potencialmente mortales, presentan una mayor resistencia o tolerancia a los antibióticos, lo que complica considerablemente la práctica clínica. Además, de manera alarmante, estudios recientes muestran que, además de la resistencia, los niveles de tolerancia de las poblaciones bacterianas, son extremadamente flexibles en un contexto evolutivo.
Las poblaciones bacterianas pueden adoptar diferentes estrategias para volverse refractarias a un tratamiento antibiótico que de otro modo sería letal. El surgimiento y diseminación de estas estrategias de supervivencia, puede ser el resultado de la transmisión vertical de mutaciones “de novo” o la transferencia horizontal de elementos genéticos móviles. Dado que la transferencia horizontal de genes (HGT) ha resultado difícil de rastrear durante la evolución del laboratorio, los parámetros ambientales que la afectan siguen siendo poco conocidos. A pesar de la importancia de la HGT, este artículo se centra principalmente en la evolución por mutaciones “de novo”, que pueden controlarse fácilmente en el laboratorio.
Una estrategia de supervivencia a los antibióticos bien documentada, es la resistencia, que suele ser conferida por cambios genéticos que permiten que las bacterias crezcan a concentraciones elevadas de fármaco. La resistencia se cuantifica de forma rutinaria mediante la concentración inhibitoria mínima (CMI), definida como la concentración de antibiótico más baja que se requiere para prevenir el crecimiento bacteriano.
Sin embargo, incluso las poblaciones con una CMI baja, pueden mostrar una supervivencia considerable cuando se enfrentan a un ataque de antibióticos. Esto se debe, en muchos casos, a la tolerancia a los antibióticos, que permite que las bacterias sobrevivan, pero no proliferen durante un tratamiento con antibióticos en dosis altas. A diferencia de los mutantes resistentes, las células tolerantes solo difieren fenotípicamente de las células susceptibles, y pueden constituir la población completa o estar presentes como una subpoblación de células.
Por otro lado, si bien la resistencia se expresa específicamente en términos de concentraciones de antibióticos, la tolerancia solo depende débilmente de la concentración aplicada. Las células tolerantes mueren mucho más lentamente que las células susceptibles en una amplia gama de concentraciones de antibióticos, lo que implica que los niveles de tolerancia pueden cuantificarse en función de la duración mínima de la muerte (MDK) de una población en concentraciones elevadas.
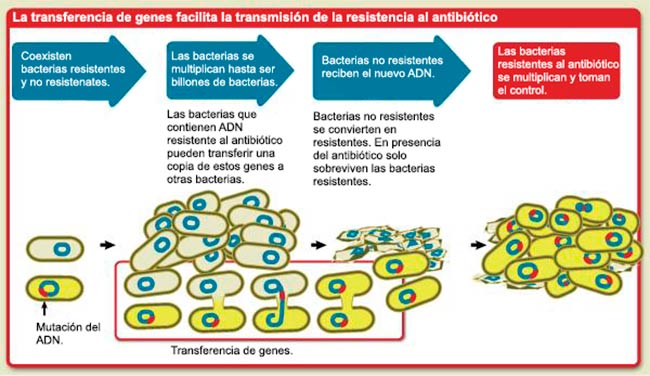
Las bacterias son conocidas por su alto potencial de adaptación cuando enfrentan diferentes tipos de estrés, incluida la terapia con antibióticos. Los cambios genéticos pueden causar un aumento de los niveles de resistencia o tolerancia, ya sea directamente, o al afectar la expresión de otros genes de resistencia o tolerancia, lo que permite a las poblaciones bacterianas hacer frente mejor a los antibióticos a los que están expuestas. Desde hace poco, esta adaptabilidad se está explorando ampliamente mediante la evolución experimental.
Así pues, a continuación, se resumen los estudios de laboratorio que brindan información sobre la evolución de la resistencia y la tolerancia, así como se explica cómo las condiciones del tratamiento podrían afectar la dirección de la evolución de las bacterias bajo antibióticos.
Evolución a alta resistencia
Como resultado de las amenazas inminentes que plantean los patógenos resistentes a los antibióticos, se ha dedicado mucha investigación a la aparición y evolución de la resistencia de bacterias bajo antibióticos. Estos estudios, a menudo se benefician de experimentos de evolución de laboratorio que imitan la adaptación de las bacterias bajo antibióticos, lo que permite la observación en tiempo real de los cambios evolutivos y la reconstrucción detallada de las trayectorias mutacionales.
La resistencia, a menudo evoluciona al exponer las bacterias a una dosis fija de antibiótico. Sin embargo, a medida que la presión de selección disminuye, una vez que se adquiere una sola mutación de resistencia, esta configuración, da como resultado, una resistencia de bajo nivel y solo confiere una visión limitada de la dinámica evolutiva de la resistencia de nivel superior. Así pues, mantener la presión de selección aumentando gradualmente la concentración de antibiótico en el espacio o en el tiempo, facilita la acumulación de mutaciones de resistencia múltiple y suele dar como resultado, mutantes altamente resistentes, imitando así la dinámica evolutiva en entornos clínicos.
La teoría predice que la selección de resistencia, es más fuerte cuando la concentración de antibiótico está por encima de la CMI, ya que estas condiciones suprimen completamente el crecimiento de células susceptibles y, por lo tanto, maximizan la ventaja de crecimiento de mutantes resistentes. Sin embargo, dado que los mutantes de un solo paso a menudo tienen sólo CMI ligeramente elevadas, es poco probable que surjan cuando la concentración de antibiótico excede un umbral llamado “concentración de prevención de mutantes” (MPC). Las concentraciones de fármaco sub-CMI, por otro lado, todavía ejercen efectos deletéreos en las células susceptibles, lo que también resulta en un fuerte enriquecimiento selectivo de mutantes altamente resistentes clínicamente relevantes.
Asimismo, la resistencia a los antibióticos, a menudo, se asocia con un costo de aptitud física, debido al mantenimiento de los mecanismos de resistencia y/o mutaciones en genes que juegan un papel esencial en el metabolismo bacteriano. Este defecto de aptitud física, generalmente, se refleja como una tasa de crecimiento reducida en condiciones sin antibióticos. En particular, un estudio reciente sugiere que los mutantes resistentes que surgen bajo estrés antibiótico subletal, exhiben defectos de aptitud más pequeños que los que surgen en dosis altas, debido a una competencia más fuerte con las células susceptibles.
Además de los numerosos experimentos de evolución in vitro, varios estudios se han centrado en la evolución de la resistencia a los antibióticos dentro de un huésped. Si bien todavía se carece de un modelo in vivo apropiado para la evolución de la resistencia, se han secuenciado grandes colecciones de aislamientos clínicos longitudinales y se han evaluado los fenotipos, lo que revela la dinámica de la evolución de la resistencia dentro del huésped, así como la transmisión de genes de resistencia de paciente a paciente.
Ha quedado claro que las tasas de aparición de resistencias in vitro, a menudo, no predicen adecuadamente el desarrollo de resistencias en entornos clínicos, debido a la participación de la HGT, distintas condiciones ambientales y tamaños de población bacteriana, costos de aptitud in vivo, interacciones de múltiples especies y otros factores que son pasados por alto en los estudios de laboratorio. Desentrañar la dinámica evolutiva de la resistencia clínica, requiere estudios longitudinales más extensos y podría estar respaldado por la recopilación sistemática de datos de sensibilidad a los antibióticos de patógenos dentro del hospedador, así como especies comensales. Los conocimientos evolutivos combinados obtenidos de los experimentos de evolución de laboratorio y los datos clínicos, son indispensables para diagnosticar y prevenir el desarrollo de resistencia a los antibióticos durante las infecciones.
Evolución a alta tolerancia
En contraste con la resistencia genética, la capacidad evolutiva de la tolerancia a los antibióticos, sólo se ha explorado recientemente. Los modelos teóricos predicen que un tratamiento periódico de altas dosis, debería favorecer inicialmente la aparición de una alta tolerancia.
Estas predicciones, se han complementado con datos de múltiples experimentos de evolución independientes, donde los científicos concluyen que es preocupante que las poblaciones bacterianas cultivadas y tratadas experimentalmente en laboratorio, albergan aproximadamente 1000 veces más células tolerantes después de solo unos pocos ciclos de tratamiento. Y aunque todavía no se han realizado experimentos de evolución similares in vivo, algunas observaciones sugieren que la evolución de la tolerancia a los antibióticos también ocurre dentro de un paciente y, por lo tanto, ya no puede pasarse por alto en la práctica clínica.
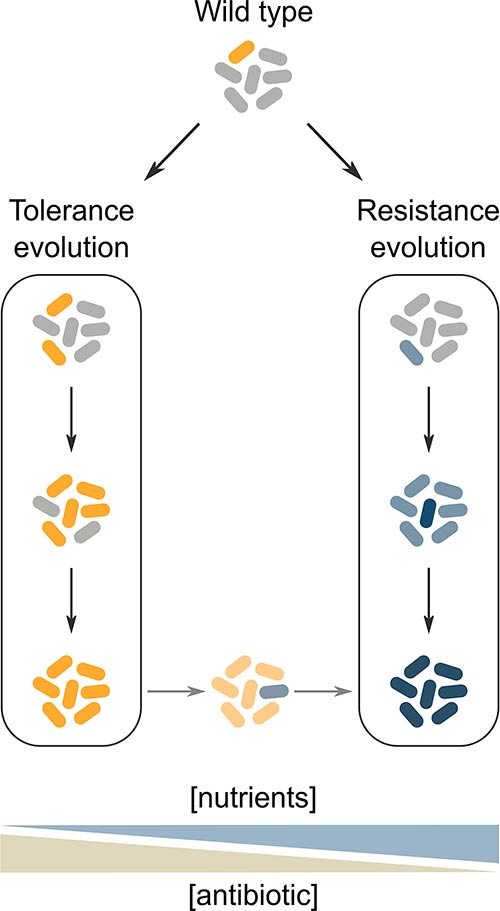
Evolución de la resistencia y la tolerancia: ¿vías alternativas a la supervivencia de bacterias bajo antibióticos?
Tanto la resistencia como la tolerancia, proporcionan una ventaja selectiva considerable a las poblaciones que están expuestas con frecuencia a los antibióticos. Sin embargo, dependiendo del programa de tratamiento con antibióticos y las condiciones ambientales, la resistencia o la tolerancia podrían ser la estrategia de supervivencia más beneficiosa. La forma en que estas condiciones afectan la dirección de la adaptación evolutiva, es un tema poco estudiado, pero se pueden inferir algunas hipótesis a partir de las propiedades de cada estrategia de supervivencia, tal y como se muestra, a continuación, en la siguiente Figura. Ésta muestra un modelo conceptual de evolución de tolerancia y resistencia en diferentes condiciones de tratamiento antibiótico. Presumiblemente, la tolerancia se ve favorecida cuando se aplican altas concentraciones de antibióticos en condiciones que restringen el crecimiento (izquierda). Por otro lado, la resistencia se ve favorecida cuando la concentración de nutrientes es lo suficientemente alta para permitir el crecimiento, mientras que la concentración de antibiótico, no debe ser demasiado alta para ser superada por mutaciones de resistencia (derecha). En condiciones que seleccionan la resistencia, una cepa tolerante desarrolla la resistencia más rápido que una cepa de tipo salvaje.
Las mutaciones de resistencia, permiten que las células crezcan durante el tratamiento con antibióticos, aunque se suelen asociar con un costo de aptitud física en condiciones sin antibióticos. Esto implica que la resistencia es, principalmente, beneficiosa para las poblaciones que están continuamente expuestas a los antibióticos. Al mismo tiempo, otros parámetros ambientales (por ejemplo, concentraciones de nutrientes), deberían ser favorables para el crecimiento, con el fin de maximizar la ventaja selectiva de los mutantes resistentes sobre las células susceptibles, que son inhibidas o muertas por el antibiótico. Como la resistencia se adquiere mediante la acumulación gradual de mutaciones, en su mayoría de bajo efecto, la selección inicial debe ocurrir en concentraciones de antibiótico suficientemente bajas para obtener la evolución.
La tolerancia, por otro lado, permite que las células sobrevivan a altas dosis de antibióticos, sin permitir el crecimiento durante el tratamiento. Por lo tanto, se espera que el aumento de la tolerancia sea la ruta evolutiva favorable cuando el tratamiento con antibióticos se produce en un entorno que no promueve el crecimiento (por ejemplo, en condiciones de limitación de nutrientes). Además, aunque las mutaciones de tolerancia también pueden estar asociadas con defectos de crecimiento en un entorno libre de antibióticos, el fenotipo tolerante a los antibióticos, en muchos casos, se expresa en condiciones inhibidoras del crecimiento. No obstante, a pesar de estos argumentos teóricos, el efecto de las concentraciones de nutrientes sobre la evolución de la tolerancia, queda por determinar experimentalmente.
Las dosis de antibióticos que son difíciles de superar por una sola mutación de resistencia, también pueden seleccionar la tolerancia, que solo depende débilmente de la concentración. La evolución de la tolerancia, requiere que dichos tratamientos de dosis alta, se interrumpan con períodos de crecimiento para permitir la división bacteriana y, por lo tanto, la selección. Si las poblaciones evolucionadas exhiben tolerancia en su conjunto o solo en una subpoblación de células, presumiblemente depende de la frecuencia del tratamiento con antibióticos. De hecho, se ha demostrado que el porcentaje de células tolerantes en poblaciones evolucionadas, se correlaciona negativamente con el intervalo de tiempo entre dos tratamientos. De manera similar, otros parámetros bacterianos, como la duración de la tolerancia en toda la población, pueden ajustarse para que coincidan con la duración del tratamiento con antibióticos.
La resistencia y la tolerancia, a menudo, se consideran estrategias de supervivencia alternativas y mutuamente excluyentes, cada una con beneficios y costos asociados a condiciones ambientales específicas. No obstante, varios estudios recientes han demostrado que la adquisición de una mutación de tolerancia no limita, sino que incluso acelera, la evolución posterior de la resistencia en condiciones favorables para ésta. Recientemente, se han hecho observaciones similares en un paciente en el que la tolerancia a un tratamiento combinado impulsó el desarrollo de resistencia. Además, se ha demostrado una interacción epistática positiva entre las mutaciones de resistencia y tolerancia, para la supervivencia bajo tratamiento con antibióticos.
Los conceptos mencionados anteriormente, podrían proporcionar una base para predecir la adaptación de bacterias bajo antibióticos en un paciente, en función de las concentraciones locales de nutrientes y de antibióticos. Sin embargo, los entornos in vivo son extremadamente complejos, heterogéneos y no están bien caracterizados, lo que complica considerablemente la extrapolación de las observaciones in vitro a las condiciones dentro del huésped. De hecho, las concentraciones locales de nutrientes y antibióticos, varían mucho entre los tejidos e incluso dentro de ellos. Además, los parámetros ambientales adicionales, como las propiedades fisicoquímicas, la compartimentación y la presencia de células inmunes, presumiblemente también desempeñan un papel en el crecimiento y la evolución in vivo de bacterias. Recientemente, se han desarrollado varias herramientas para caracterizar el entorno local que es detectado por las bacterias durante una infección, que pueden imitarse en experimentos de evolución de laboratorio.
Además, la mayoría de los estudios de laboratorio, se centran en tratamientos con un solo fármaco, mientras que los pacientes en la clínica suelen ser tratados con combinaciones de antibióticos. Un estudio reciente, mostró que los tratamientos combinados afectan fuertemente la evolución de la tolerancia y la resistencia, destacando así la necesidad de cerrar esta brecha.
En general, la investigación futura debe dirigirse a la evolución in vivo para reconstruir las trayectorias evolutivas del mundo real e identificar los parámetros ambientales que afectan la dirección de la evolución.
Fuente de este artículo
Este artículo fue publicado originalmente en la revista Plos Pathogens, bajo la referencia: Windels EM, Van den Bergh B, Michiels J (2020). Bacteria under antibiotic attack: Different strategies for evolutionary adaptation. PLoS Pathog 16(5): e1008431. https://journals.plos.org/plospathogens/article?id=10.1371/journal.ppat.1008431; y el cual está disponible con acceso libre en la propia web de Plos Pathogens.


