En este artículo, elaborado por dos científicas del CSIC, se explica cómo y dónde tiene lugar la morfogénesis de adenovirus, concluyendo que el ensamblaje y el empaquetamiento se producen simultáneamente en la periferia del centro de replicación.
Presentación
“Localization of adenovirus morphogenesis players, together with visualization of assembly intermediates and failed products, favor a model where assembly and packaging occur concurrently at the periphery of the replication center” (“La localización de los componentes de morfogénesis de adenovirus, junto con la visualización de intermediarios de ensamblaje y productos fallidos, favorecen un modelo en el que el ensamblaje y el empaquetamiento se producen simultáneamente en la periferia del centro de replicación”), es un artículo publicado en la revista “PLOS Pathogens” en abril de 2017 por las científicas Gabriela N. Condezo y Carmen San Martín del CSIC de Madrid (España), en el que explican el descubrimiento de la localización de la morfogénesis de adenovirus, lo cual puede ayudar a diseñar antivirales para tratar enfermedades producidas por éstos, así como también puede permitir desarrollar virus que sirvan como herramientas terapéuticas.
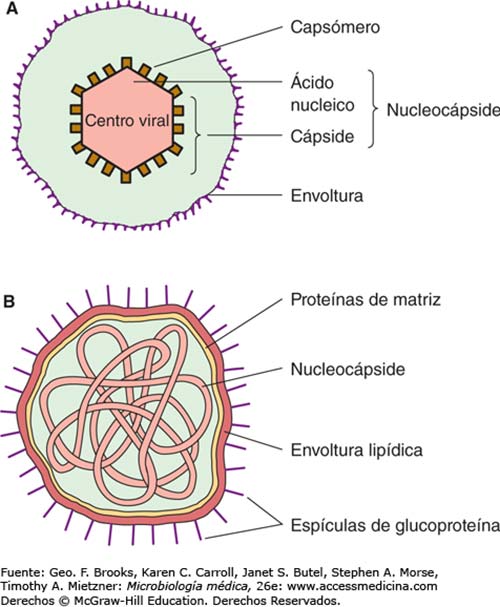
Los adenovirus (Adenoviridae) son un grupo de virus que infectan tanto a humanos como animales. Son virus no encapsulados (sin envoltura que rodea la cápside vírica) con ADN bicatenario, que pueden causar infecciones en las vías respiratorias, conjuntivitis, cistitis hemorrágica y gastroenteritis.
Los adenovirus pueden causar problemas clínicos graves en personas inmunodeprimidas, pero también pueden aprovecharse para pasar de ser un patógeno a ser una herramienta terapéutica útil. Por tanto, comprender su biología es crucial para tener éxito en la curación de las infecciones por adenovirus y para reutilizar el virus para nuestra ventaja.
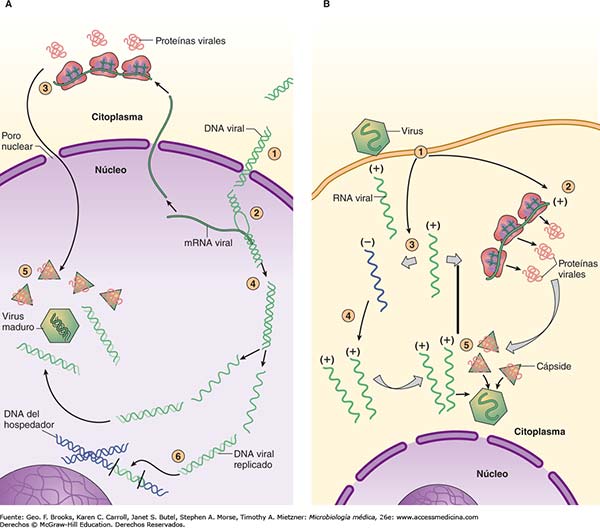
El ensamblaje de adenovirus se produce en lugares específicos de las células infectadas, donde proteínas de la cubierta principal, junto con proteínas de la cubierta secundaria y la proteína de empaquetamiento L1 52/55 kDa, se ensamblan en cápsides vacías. Asimismo, los genomas virales y las proteínas del núcleo se insertan también en estas cápsides para producir partículas inmaduras no infecciosas denominadas viriones. Éstos contienen la versión precursora de varias proteínas de la cápside (pIIIa, pVI, pVIII) y del núcleo (pVII, pμ, pTP), así como la L1 52/55 kDa. Los viriones maduros se producen después de la escisión de estos precursores por los adenovirus.
La cápside vírica que contiene y protege el genoma de los adenovirus, está formada por más de diez tipos de proteínas que deben fabricarse y ensamblarse de forma muy coordinada. Dicho ensamblaje y la introducción del genoma en su interior, debe ser preciso y correcto para que el virus pueda propagarse. Para introducir el genoma en la cápside existen dos tipos de mecanismos: concertado, en el que la cápsula de proteínas es construida en torno al genoma, o secuencial, donde primero se ensambla una cápside vacía con un canal, por el que luego se introduce el genoma.
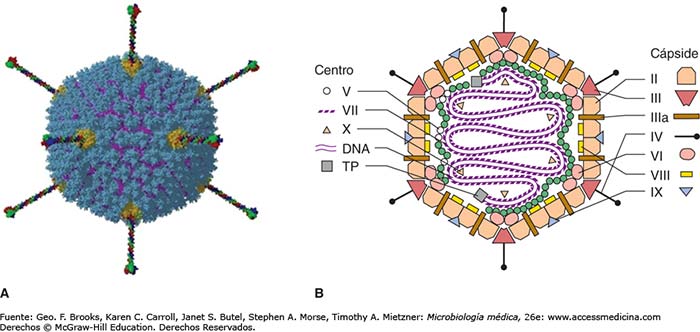
La morfogénesis de adenovirus es un proceso complejo y a pesar de que se llevan estudiando los adenovirus durante más de 60 años, hasta ahora todavía no se sabía dónde se realiza el ensamblaje ni mediante qué mecanismo se hacía. En este trabajo, sus autoras abordan estas dos preguntas, determinando la ubicación en el núcleo celular donde se reúnen los factores de ensamblaje y empaquetado de adenovirus, describiendo productos de ensamblaje fallidos que contienen genomas desempaquetados, y mostrando por primera vez imágenes de fragmentos de cápside de adenovirus en el proceso de introducir el ADN viral.
Métodos y Resultados
En este trabajo han empleado técnicas de inmunofluorescencia e inmunomicroscopía electrónica para rastrear los factores de empaquetamiento y las proteínas estructurales en los últimos tiempos después de la infección por adenovirus, ya sea por el virus de tipo salvaje o un mutante, lo cual ha permitido determinar la localización de la factoría viral de la morfogénesis de adenovirus. Los patrones de etiquetado de estos factores y las modificaciones nucleares inducidas por Ad5 y el mutante de empaquetamiento diferido Ad5/FC31, se comparan para obtener nueva información sobre la conexión entre el ensamblaje y el envasado. Asimismo, se muestra que los representantes de todos los factores de ensamblaje están presentes en la zona replicativa periférica (PRZ), la cual constituye el lugar de la morfogénesis de adenovirus.
Además, en este estudio se muestran, por primera vez, imágenes con fragmentos de cápsides de distintos tamaños unidas a estructuras formadas por el genoma del virus y unas proteínas que ayudan a condensarlo para que quepa dentro de la cápside. Estas imágenes apoyan el modelo de ensamblaje concertado más que el secuencial.
Asimismo, los productos intermediarios de ensamblaje y abortivos observados, también favorecen un modelo de ensamblaje y envasado concertado que comprende dos vías: una para proteínas de cápside y otra para componentes centrales, y solo cuando ambas vías se acoplan por la interacción correcta entre las proteínas de empaquetamiento y el genoma, se produce la partícula viral. De hecho, se ha visto que el desacoplamiento genera la acumulación de cápsides vacías y núcleos sin empaquetar.
El empaquetamiento de adenovirus comienza desde el extremo izquierdo del genoma, donde se encuentra la secuencia de empaquetamiento específica C. Se requieren varias proteínas codificadas por virus para que se produzca el empaquetamiento del genoma: IVa2, L1 52/55 kDa, L4 22 kDa, L4 33 kDa y IIIa. En concreto, IIIa interactúa con L1 52/55 kDa in vitro y con C in vivo, lo que indica cómo el genoma puede unirse a la cápside durante el ensamblaje. La evidencia disponible sugiere que L1 52/55 kDa media la asociación estable entre el ADN viral y la cápside vacía para producir una partícula completa. Así, la producción de cápsides vacías por mutantes termosensibles o de deleción, demuestra que estas proteínas son necesarias para el empaquetamiento del ADN pero no para el ensamblaje de la cápside.
Sin embargo, las proteínas IVa2 y L1 52/55K se encuentran tanto en cápsides vacías como unidas a C, lo que sugiere que pueden estar presentes en dos grupos separados: uno asociado a cápsides preparadas para recibir ADN viral para encapsular, y un segundo grupo unido a C para promover la interacción entre el ADN viral y los componentes de la cápside.
Por otra parte, se sugiere que el ensamblaje de adenovirus se combina con la síntesis de ADN y esto favorece también el modelo concertado. De hecho, la inhibición de la síntesis de ADN, pero no de las proteínas virales, da como resultado una reducción del ensamblaje del virus, a pesar de la presencia de ADN viral acumulado previamente a la inhibición; de modo que solo el ADN que se sintetiza se empaqueta en viriones maduros.
A continuación se describen los métodos de trabajo llevados a cabo en este trabajo.
Células, virus y anticuerpos
Se cultivaron células HEK293 (ATCC CRL-1573) a 37°C en medio de Eagle modificado por Dulbecco, suplementado con suero bovino fetal al 2%, 10 unidades 10μg/ml de penicilina-estreptomicina, 0,05mg/ml de gentamicina, 4mM de L-Glutamina y 1X de solución de aminoácido no esencial.
El virus control de tipo salvaje fue la variante Ad5 Ad5GL, en la que la región E1 ha sido eliminada y sustituida por GFP y genes de luciferasa de luciérnaga. La variante Ad5/FC31, por su parte, tiene una inserción attB/attP flanqueando C y una cassette GFP después de C. Posteriormente se llevó a cabo la propagación y purificación del virus.
Los anticuerpos primarios utilizados fueron: monoclonal abcam de la rata anti BrdU, suero de rata anti Ad5 pVII, ratón anti Ad2 IVa2 suero, ratón anti Ad5 DBP monoclonal, suero de conejo anti Ad5 L1 52/55 kDa, y suero de fibra de conejo anti Ad5; mientras que los anticuerpos secundarios usados fueron: para ensayos de inmunofluorescencia, Alexa Fluor594 Goat Anti-Rat, Alexa Fluor555 Goat Anti-Rat, Pacific Blue Goat Anti-Mouse y Anti-Conejo de Cabra Azul del Pacífico; y para inmunoEM, 10nm de oro conjugado anti-conejo, 10nm de oro conjugado anti-ratón, y 15nm de oro conjugado anti-rata.
Microscopía electrónica convencional de células infectadas
Las células HEK293 cultivadas en placas de cultivo p100 hasta una confluencia del 70%, se infectaron con Ad5/FC31 o Ad5 wt con una multiplicidad de infección (MOI) de 5. En el momento deseado, después de la infección, el medio se eliminó y las células se fijaron con un 2% glutaraldehído y 1% de ácido tánico en 0.4MHEPES pH 7.2 durante 1.5h a temperatura ambiente. Se recogieron secciones ultrafinas (~70nm) en rejillas de níquel recubiertas con Formvar, se tiñeron con acetato de uranilo saturado y citrato de plomo, y se examinaron en un microscopio electrónico de transmisión JEOL JEM 1230 a 100kV. Las células infectadas falsas se procesaron de la misma manera que un control.

Microscopía de inmunofluorescencia
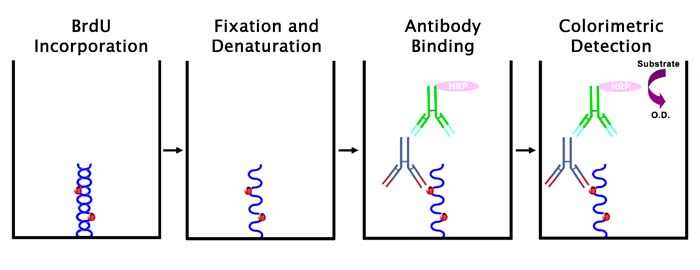
Las células monocapas HEK293 cultivadas en copas se infectaron con Ad5 wt o Ad5/FC31. La infección se sincronizó incubando las células durante 30 minutos a 4ºC y luego durante 30 minutos a 37ºC. Luego, se retiraron los inóculos y se añadió medio. Se llevó a cabo el marcado BrdU modificando en algunos aspectos el protocolo a seguir. Las muestras se incubaron con anticuerpo primario en saponina al 0,5% y FBS al 2% en PBS durante 45 minutos. Después se realizó la incubación con anticuerpos secundarios diluidos en saponina al 0,5% y FBS al 2% en PBS en oscuridad. Y después de un enjuague final con PBS, se montaron los cubreobjetos en portaobjetos de vidrio usando ProLong.
Las imágenes se tomaron usando un sistema confocal multiespectral Leica TCS SP5 y los análisis de estas imágenes se llevaron a cabo con Image J.
Los controles negativos consistieron en incubaciones sin anticuerpo primario e inmunomarcación de células infectadas simuladas. En experimentos de marcado doble, la ausencia de reactividad cruzada se verificó mediante incubaciones con solo uno de los anticuerpos primarios y el anticuerpo secundario correspondiente al otro.
Sustitución de congelación y microscopía de inmunoelectrones
El marcado de BrdU del ADN recién sintetizado se llevó a cabo como se describe para inmunofluorescencia, con células HEK293 cultivadas en placas de cultivo p100 en lugar de cubiertas. Las células infectadas y de control se fijaron con paraformaldehído al 4% en PBS. Después de enjuagar tres veces con PBS, se añadió glicerol gota a gota hasta el 15% de concentración final. Después de 15 minutos a 4ºC, se aumentó el glicerol al 30% y 15 minutos más tarde, las células se recogieron, sedimentaron y congelaron mediante congelación por inmersión en etanol líquido usando un émbolo Leica CPC.
La sustitución de congelación se llevó a cabo en un sistema automático de sustitución de congelación (AFS) Leica EM. Las muestras se mantuvieron en acetato de uranilo al 0,5% en metanol seco durante 60 horas a -90°C, con varios cambios de solución deshidratante. Luego, la temperatura se elevó de manera controlada para alcanzar -40°C después de 7h y se mantuvo hasta el final del procedimiento. Las muestras se enjuagaron con metanol seco y se infiltraron con concentraciones crecientes de Lowicryl HM20 en metanol durante 24h. La polimerización se llevó a cabo mediante irradiación UV durante 48h a -40°C, y luego 48h a 20°C. Se obtuvieron secciones ultrafinas, como se describe en la sección de microscopía electrónica convencional.

Para los ensayos inmunoEM, las rejillas que llevan secciones ultrafinas sustituidas con congelación se colocaron en TBG durante 10min, se incubaron con el anticuerpo primario en TBG durante 30min, se lavaron 3 veces con PBS, flotando en 4 gotas de TBG (5 min por gota) y se incubaron en anticuerpos secundarios conjugados con oro diluidos en TBG durante 30 minutos. Luego, las rejillas se lavaron 3 veces con PBS y agua milli-Q, y se tiñeron con acetato de uranilo saturado. Todas las incubaciones se llevaron a cabo a temperatura ambiente. Los controles negativos consistieron en incubaciones sin anticuerpo primario e inmunomarcación de células infectadas simuladas.
Para el etiquetado anti-BrdU, se requirió un paso adicional. Las secciones se trataron con 0,2mg/ml de proteinasa K durante 15 min a 37°C, luego se lavaron con agua milli-Q y se desnaturalizaron con HCl 2N durante 25 min. Después de varios enjuagues en agua milli-Q, el protocolo continuó con la incubación TBG.
Para desenmascarar la proteína VII, las secciones se hicieron flotar en tres gotas de tampón de ADNasa (5 min por gota), luego se incubaron con 50μg/ml de ADNasa durante 1 hora a 37°C, se enjuagaron en agua milli-Q y se transfirieron a TBG para iniciar el protocolo de etiquetado de inmunoanticrepano. La incubación con el anticuerpo primario anti-VII se realizó durante la noche a 4ºC.
Y tras describir los métodos, se narran ahora los resultados hallados en los distintos métodos.
Localización por inmunofluorescencia de los factores de empaquetamiento del genoma de adenovirus
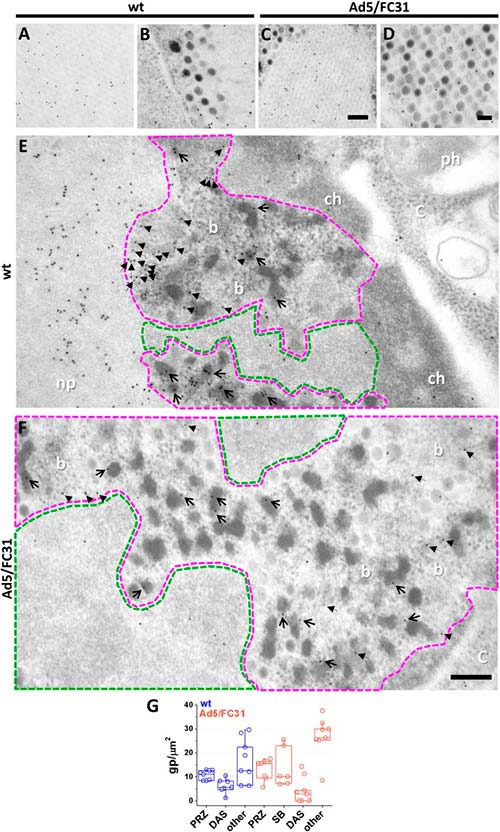
Para comenzar a definir la región nuclear donde ocurre el empaquetamiento de adenovirus, los genomas virales y los factores de empaquetamiento IVa2 y L1 52/55 kDa se localizaron mediante microscopía de inmunofluorescencia en células Ad5 wt o Ad5/FC31 infectadas en los últimos tiempos después de la infección.
BrdU presentó un patrón de anillo difuso y se detectó más ADN (más células marcadas) en Ad5/FC31, aunque las mediciones en vistas ortogonales, indicaron que los anillos marcados eran en realidad elipsoides. Dado que este patrón de etiqueta es similar al anteriormente informado para focos de replicación, se supuso que la etiqueta BrdU del anillo difuso correspondía a la PRZ (Zona de Replicación Periférica) y que el área no etiquetada dentro del anillo podía corresponder a la DAS.
Para investigar dónde se encuentran las proteínas de empaquetamiento y los genomas virales, se llevaron a cabo ensayos de doble etiquetado para BrdU y L1 52/55kDa, o BrdU y IVa2. La etiqueta para IVa2 fue débil, con lo que tal vez se correlaciona con el bajo número de copias de la proteína en los viriones o con una reactividad de anticuerpos limitada. L1 52/55kDa, por su parte, se detectó en la periferia de los centros de replicación marcados con BrdU, donde ambas señales se entremezclaron. No hubo etiqueta para L1 52/55kDa en las áreas sin etiqueta de BrdU correspondientes al DAS. La presencia de L1 52/55kDa y genomas virales en la PRZ sugiere que el empaquetamiento de ADN ocurre en esta área.
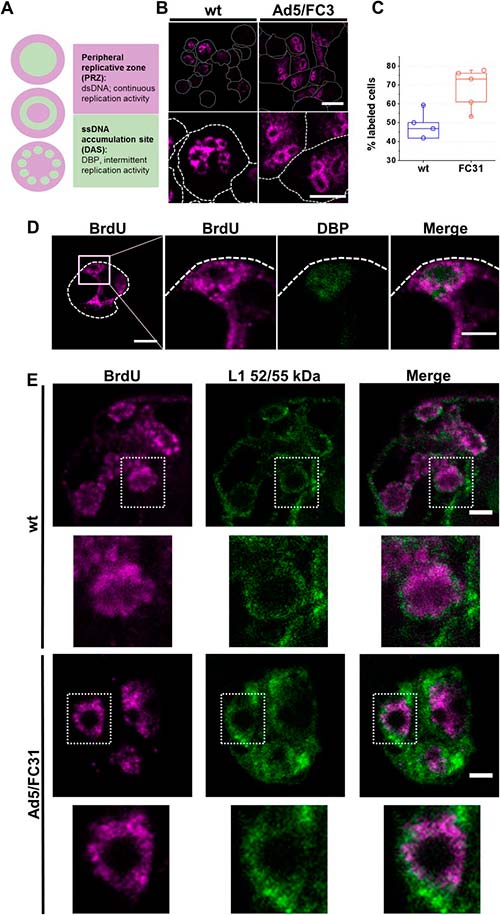
Localización por microscopía inmunoelectrónica de factores de empaquetamiento del genoma de adenovirus
Para corroborar la identidad de las posibles PRZ, las células infectadas se trataron con BrdU y se procesaron para inmunoEM mediante la sustitución de congelación (FS), para preservar tanto la estructura como la inmunorreactividad. En las muestras de FS, las posibles zonas PRZ y DAS se identificaron en función de su densidad de electrones y la presencia de EOG y partículas virales.
La etiqueta para BrdU se encontró específicamente en la posible zona PRZ, confirmando su identidad y se asoció frecuentemente a partículas completas (viriones), EOG y material suelto con electrones densos, lo que confirma que contienen ADN viral. Los diferentes niveles de densidad de electrones de estas estructuras sugieren diferentes grados de condensación de ADN, yendo desde la mayoría relajados (haces) hasta los más condensados (viriones y EOG).
Por su parte, el área de electrones claros propuesta para ser el DAS, mostró una etiqueta débil, de acuerdo con los resultados de inmunofluorescencia y los informes previos que indican baja actividad replicativa en esta región.
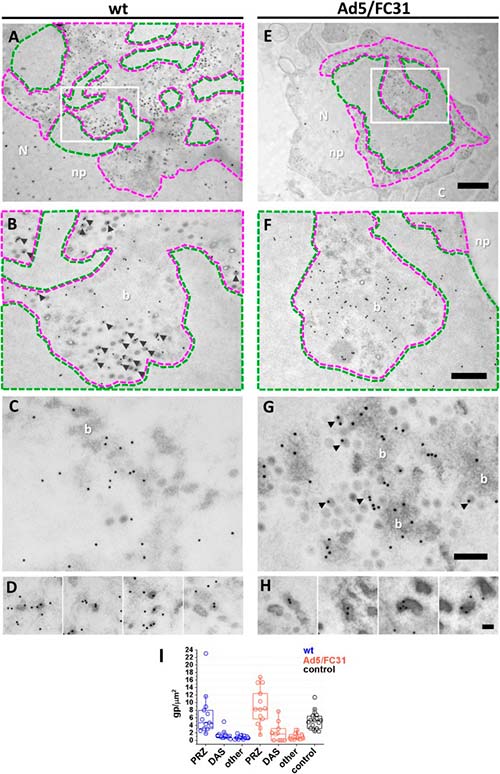
A continuación, se analizó la localización de las proteínas de empaquetamiento. Se observó una etiqueta dispersa para L1 52/55 kDa en todos los núcleos infectados, incluida la PRZ. Sin embargo, se observaron muy pocas partículas de oro en el DAS, lo que respalda la especificidad de la etiqueta. La señal de L1 52/55 kDa en la PRZ se asoció a las características zonas electrón-densas. Como se esperaba, L1 52/55 kDa se detectó en partículas virales, particularmente en aquellas con menor densidad de electrones que indicaban un empaquetamiento incompleto.
La etiqueta en partículas virales a menudo presentó un patrón de arco con un caparazón, lo que indicaba la presencia de esta proteína dentro de la cápside. Los patrones de arco también se encontraron en EOG, sugiriendo la formación de un caparazón de L1 52/55 kDa en el material electrón-denso que contienen. Estas observaciones son consistentes con la presencia de L1 52/55 kDa en dos grupos: uno que se une al ADN viral (paquetes y EOG), y otro que se une a proteínas de la cápside (cápsides electrón-claras). Además, también se vio que grupos de partículas de oro también formaban pequeños arcos y se encontraron con frecuencia cerca de la PRZ, sugiriendo fragmentos de caparazón L1 52/55kDa en su camino para ensamblarse con genomas virales o proteínas de la cápside.
La etiqueta para la proteína IVa2 resultó débil, tal y como se observó previamente en la inmunofluorescencia. Sin embargo, la señal para IVa2 estaba presente en la PRZ en material denso en electrones (haces y algunos EOG).
En resumen, la inmunoEM confirmó que los genomas de adenovirus y las proteínas de empaquetamiento están presentes en la PRZ, la cual también contiene partículas virales. Estos resultados sugieren que la PRZ podría ser la ubicación en el núcleo donde tiene lugar la encapsulación del genoma en la morfogénesis de adenovirus.
Localización de proteínas estructurales de adenovirus mediante microscopía inmunoelectrónica
Para evaluar mejor la hipótesis de que el ensamblaje de adenovirus se produce en la PRZ, se analizó la presencia de proteínas del núcleo (VII) y de la cápside (fibra). La etiqueta para la proteína VII se observó exclusivamente en la PRZ, con frecuencia en EOG y en cantidades inferiores en haces de ADN, lo que corrobora la idea de que contienen ADN viral condensado en diferentes grados por las proteínas del núcleo. Además, la proteína VII también se detectó en partículas virales.
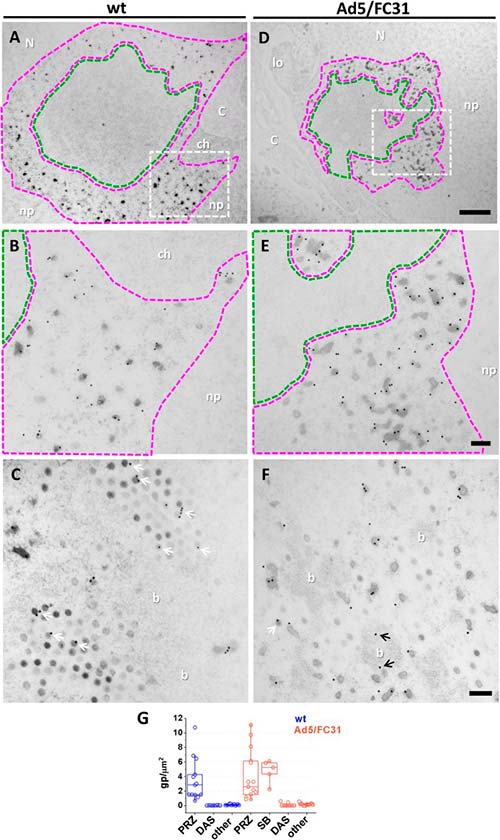
El análisis de la localización de la proteína de la cápside en células infectadas con adenovirus no resultó sencillo, ya que se producen en gran exceso. Se detectaron anticuerpos contra cristales de proteína marcados en fibra y partículas virales, como se esperaba. La fibra también se detectó en la PRZ y en EOG y haces, pero solo se observó una señal débil en el DAS, lo que respalda la especificidad de la etiqueta. La presencia de proteínas del núcleo y de la cápside, junto con el ADN vírico y factores de empaquetamiento, así como partículas virales, indica que el PRZ es el sitio de ensamblaje de adenovirus, es decir, el lugar de la morfogénesis del adenovirus.
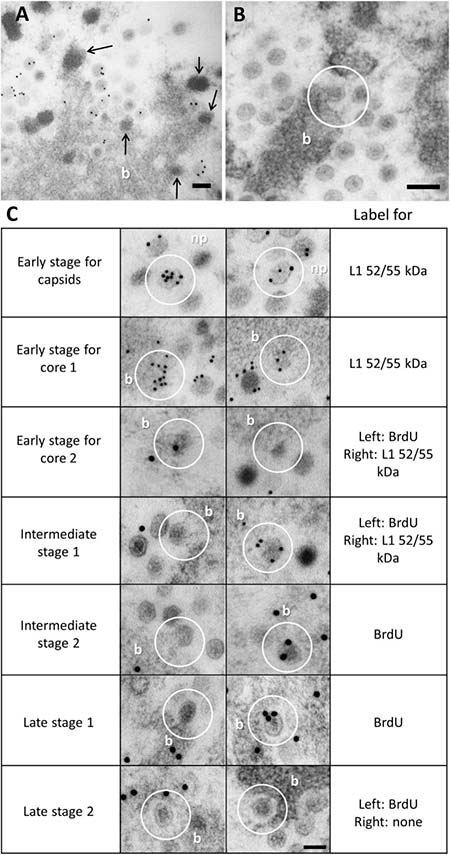
Compuestos intermediarios de adenovirus
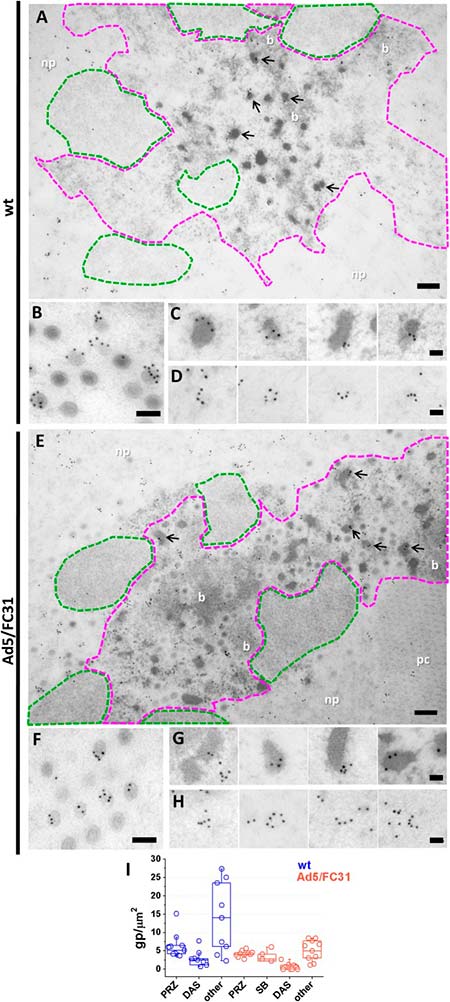
Después de determinar que los representantes de todos los componentes de la morfogénesis de adenovirus (genoma, factores de empaquetamiento, proteínas del núcleo y de la cápside) estaban presentes en la PRZ, los autores del presente estudio abordaron la cuestión sobre dónde exactamente se estaba produciendo el ensamblaje dentro de esta región.
Tanto los EOG como los haces fueron positivos para todos los factores de ensamblaje probados, y tanto las partículas virales como los EOG se encontraron a menudo en la periferia del haz de ADN, lo que sugiere una relación topológica. La observación detallada de las muestras de FS reveló la mitad de las cápsides que envuelven las condensaciones de ADN sobresaliendo de los haces, lo que indica que éstos son muy probablemente los sitios de ensamblaje. Sin embargo, los intermediarios de ensamblaje fueron extremadamente infrecuentes en las infecciones por Ad5 wt y muy difíciles de encontrar en Ad5/FC31, lo que indica que el ensamblaje de adenovirus es un proceso altamente cooperativo.

El examen exhaustivo de todas las muestras de FS produjo una posible secuencia de eventos en el ensamblaje de adenovirus. Por un lado, los fragmentos de la cápside se ensamblan conteniendo L1 52/55 kD, y por otro lado, L1 52/55 kDa también aparece en la periferia de los haces de ADN, a menudo en pequeñas protrusiones electrón-densas que sugieren la acción condensadora de las proteínas del núcleo. Por tanto, se propone que estas protuberancias son núcleos virales nacientes, que contienen uno de los dos grupos L1 de 52/55 kDa (el que está unido a la secuencia de empaquetamiento) y que sirven como punto de reclutamiento para el otro grupo L1 de 52/55 kDa (el único límite a los fragmentos de la cápside).
Los fragmentos entrantes de la cápside se ensamblan, por tanto, alrededor del núcleo coalescente y crecen gradualmente hasta que se forma la partícula completa y finalmente, se desprende del paquete de ADN. Aunque las imágenes EM son instantáneas estáticas, la observación por primera vez de cápsides de adenovirus que se ensamblan alrededor de núcleos nacientes, sugiere un mecanismo de ensamblaje y empaquetado concertado en lugar de secuencial.
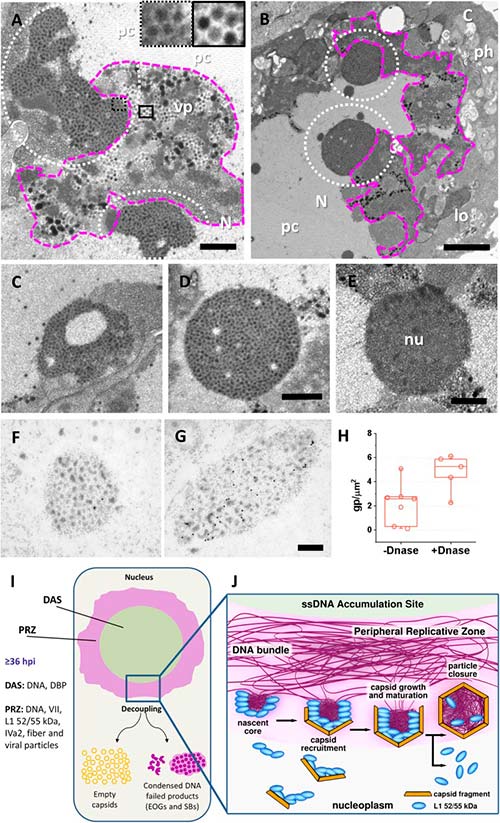
Productos de ensamblaje fallidos de la morfogénesis de adenovirus
Las cápsidas vacías generadas por mutantes de adenovirus con defectos de empaquetamiento son productos de ensamblaje defectuosos fabricados por los componentes de la cápside. Sin embargo, no se conoce el destino de los componentes del núcleo no utilizados en los mutantes defectuosos de empaquetamiento.
Aquí se muestra que los EOG surgen de haces de ADN virales y están etiquetados para factores estructurales y de empaquetamiento, pero adoptan formas y tamaños variables, diferentes de los esperados para una partícula viral. Por lo tanto, los EOG se pueden interpretar como una clase diferente de eventos de ensamblaje fallidos: núcleos cuya asociación con fragmentos de cápside no tuvo éxito. De acuerdo con esta hipótesis, se observó que EOG fueron más abundantes en Ad5/FC31 que en infecciones wt.
Además de la cantidad de EOG y partículas de luz, la diferencia más notable entre las células infectadas Ad5/FC31 y wt fue una nueva estructura que consta de puntos con una densidad de electrones muy alta, incrustada en un fondo de alta densidad de electrones, que los autores denominaron «cuerpo pelado» (SB) debido a su apariencia. Además, se descartó la posibilidad de que los SB fueran nucléolos compactos, ya que las motas son mayores que los puntos de los nucléolos y de tamaño similar a las virales.
Los SB se observaron en células infectadas Ad5/FC31 y su presencia fue más notable a las 48h después de la infección, cuando tenían una organización lobular y suelta, mientras que en los últimos tiempos parecían más compactos y circulares. La búsqueda exhaustiva reveló que los SB también estaban presentes en las células infectadas Ad5 wt, pero su aparición fue extremadamente escasa: solo se encontraron 2 SB en una muestra de 36 células infectadas Ad5 wt, mientras que se hallaron 14 SB en 45 células infectadas Ad5/FC31, dando un 5.5% vs 31.1% de probabilidad de encontrar un SB en una célula infectada. Debido a su tamaño y textura, las motas en los SB son una reminiscencia de núcleos virales, los cuales, a menudo se encontraron adyacentes a la PRZ. Por tanto, se planteó la hipótesis de que los SB podrían ser regiones de PRZ que contienen genomas virales condensados que no se han empaquetado debido a la mutación Ad5/FC31.
Para evaluar dicha hipótesis de que los SB contienen genomas de adenovirus condensados, se probó la presencia de dos componentes principales (ADN y proteína VII). En los experimentos iniciales de inmunomarcación, no se observó señal para BrdU (ADN viral) y la señal para VII fue baja. Éstos últimos se trataron con ADNasa antes de la incubación con el anticuerpo anti-VII, lo cual aumentó la señal para VII e indicó, por tanto, que no solo los SB contienen proteína VII, sino que también contienen ADN y, por lo tanto, núcleos virales.
Algunos SB tenían forma de anillo con un centro de borrado de electrones con reminiscencias del DAS, sin embargo, no se encontraron partículas virales (ni vacías ni llenas) en ellos. Por tanto, se concluye que los SB son PRZ tempranas, donde comenzó el reclutamiento de cápside a núcleo, pero fallaron debido a la mutación Ad5/FC31, la cual interfiere con la interacción entre las proteínas de empaquetamiento y C, provocando que el ensamblaje no pueda continuar.
Discusión
El empaquetamiento de adenovirus (genoma, IVa2, L1 52/55k) y los factores de ensamblaje (proteínas del núcleo y de la cápside) se encuentran en la PRZ, donde también se hallan partículas de virus. Es importante destacar que los genomas virales y la proteína VII del núcleo se localizaron exclusivamente en la PRZ. Estas observaciones indican que la PRZ es la fábrica de montaje de adenovirus y no solo la zona de replicación de ADN, como se había descrito. Esta localización del sitio de ensamblaje es consistente con la evidencia previa que indica que la replicación y el ensamblaje están acoplados y por tanto, deberían ocurrir en el mismo lugar. Además, las imágenes de EM de intermediarios de ensamblaje en células infectadas que muestran cápsides que crecen alrededor de núcleos, proporcionan una nueva evidencia para respaldar el modelo de ensamblaje concertado.
Por tanto, en este trabajo se propone el siguiente modelo para la morfogénesis de adenovirus. Por un lado, las proteínas de empaquetamiento se unen a C en los haces de ADN producidos durante la replicación del genoma. En la periferia de los haces, los genomas virales comienzan a condensarse por acción de las proteínas del núcleo. Por otro lado, en áreas cercanas a la PRZ, L1 52/55k se une a IIIa en fragmentos del caparazón icosaédrico. Los dos grupos de L1 52/55 kDa (en fragmentos de cápside y núcleos nacientes), interactúan y actúan como un velcro para reclutar y atar el núcleo condensado a la cápside naciente. El crecimiento de la cápside se produce mediante la adición de capsómeros u otros fragmentos alrededor del núcleo, mientras que simultáneamente L1 52/55k se escinde por AVP y se elimina de la partícula antes del cierre de la cápside y la retirada del haz de ADN.
Los cambios en C o en las proteínas de empaquetamiento que deterioran su interacción mutua, obstaculizarían estos procesos en diferentes puntos, resultando en partículas sin genoma con diferentes grados de ruptura de maduración y núcleos abandonados que finalmente, se fusionarían en EOG y SB.
Además, las observaciones aquí indicadas y el modelo de ensamblaje propuesto, coinciden notablemente con predicciones computacionales recientes sobre el ensamblaje de depósitos proteicos alrededor de una carga multiparticulada simultáneamente coalescente, que en este caso sería el núcleo organizado en unidades de proteínas de ADN similares a nucleosomas.
Cabe indicar que se requiere un delicado equilibrio entre las interacciones caparazón-caparazón, carga-carga y caparazón-carga, para que ocurra el ensamblaje correcto de partículas completas. Las mutaciones que deterioran la red de interacciones, alteran este equilibrio y conducen a productos de ensamblaje abortivos. En particular, los resultados de este estudio indican que, cuando el acoplamiento del ensamblaje de cápsides-núcleo es correcto, la morfogénesis de adenovirus es un proceso rápido y altamente cooperativo, como lo indica la escasez de intermediarios de ensamblaje en las dos variantes de virus estudiadas, pero más notablemente en Ad5 wt. La mutación Ad5/FC31 cambia las interacciones caparazón-núcleo, lo que permite capturar los intermediarios de ensamblaje en un proceso que, de otro modo, sería demasiado rápido para ser observado experimentalmente.
En resumen, en este trabajo se presenta la evidencia que define la ubicación de la fábrica de ensamblaje de adenovirus, lo que favorece un mecanismo de ensamblaje y empaquetado concertado para la morfogénesis de adenovirus. Sin embargo, aún quedan por responder numerosas preguntas para comprender completamente el mecanismo de ensamblaje de adenovirus.
Una de ellas es si sería necesario un contacto inicial, aunque imperfecto, entre la cápside y el núcleo (a través de las proteínas de empaquetamiento) para que las cápsides comiencen a ensamblarse. La existencia de numerosos mutantes de proteínas de empaquetamiento que producen grandes cantidades de cápsides vacías indicaría que este no es el caso. Sin embargo, un mutante DBP termocondicional con una reducción de 3 veces en la replicación del genoma no produjo ningún tipo de partículas, y las partículas incompletas de Ad5 /FC31 habían iniciado al menos el empaquetamiento. Además, dado que una variedad de proteínas (L1 52/55 kDa, IVa2, L4 33 kDa, IIIa) tienen un papel en la encapsulación, es posible que la eliminación de solo una de ellas, permita llevar a cabo las etapas iniciales de empaquetado y, por lo tanto, el ensamblaje de la cápside.
Por otro lado, el papel de la secuencia de empaquetamiento C también es intrigante. La caracterización molecular y estructural sistemática de los productos de ensamblaje de los diversos mutantes sería necesaria para evaluar este punto. Y otra pregunta sin respuesta es si el primer fragmento de la cápside que se reclutará en el núcleo es elegido al azar o se define por la presencia de alguna «característica especial”, como podría ser la presencia del empaque putativo ATPasa IVa2.
Fuente publicación
Este artículo fue publicado originalmente en la revista “PLOS Pathogens” bajo la referencia: Condezo, G. N., & San Martín, C. (2017). Localization of adenovirus morphogenesis players, together with visualization of assembly intermediates and failed products, favor a model where assembly and packaging occur concurrently at the periphery of the replication center. PLoS Pathogens, 13(4), e1006320. doi.org/10.1371/journal.ppat.1006320; estando disponible con acceso libre en la web de NCBI.
