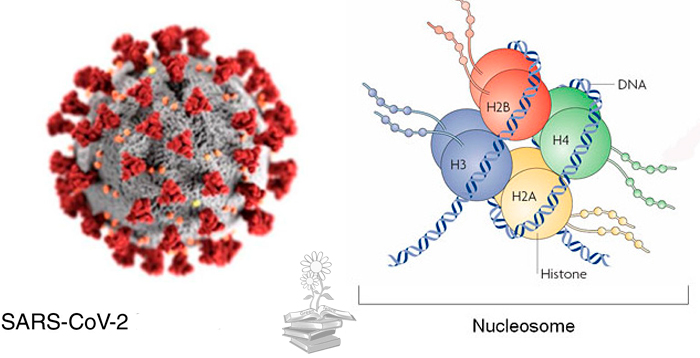
Algunas veces, las proteínas virales amortiguan las respuestas antivirales al imitar regiones críticas de las proteínas histonas humanas. Por otra parte, hay estudios que demuestran que el SARS-CoV-2 altera notablemente la regulación epigenética de las células hospedadoras, pero, sin embargo, no se sabe cómo. Por tanto, los autores de este artículo científico han conseguido demostrar que el SARS-CoV-2 altera la regulación epigenética de la célula hospedadora mediante mimetismo de histonas.
Presentación
“SARS-CoV-2 disrupts host epigenetic regulation via histone mimicry” (“El SARS-CoV-2 altera la regulación epigenética del hospedador a través de la imitación de histonas”), es un artículo científico elaborado por Kee, J., Thdium, S., Renner, D., Palozola, K., Zhang, Z., Li, Y., Lan, Y., Cesare, J., Poleshko, A., Kiseleva, A., Truitt, R., Cardenas-Diaz, F., Zhang, X., Xie, X., Kotton, D., Alysandratos, K., Epstein, J., Shi, P., Yang, W., Morrisey, E., García, B., Berger, S., Weiss, S. y Krob, E.; y publicado en Octubre de 2022 en la revista Nature (volumen 610), en el que se demuestra que el SARS-CoV-2 (causante del COVID-19) altera la regulación epigenética de la célula hospedadora mediante mimetismo de histonas, lo cual contribuye a la gravedad de la enfermedad.
En parte debido a su capacidad para suprimir eficazmente las respuestas de las células hospedadoras, en algunos casos, las proteínas virales amortiguan las respuestas antivirales al imitar regiones críticas de las proteínas histonas humanas, las cuales envuelven el ADN en estructuras complejas y controlan el acceso al genoma. Por tanto, el mimetismo de las histonas permite que los virus alteren la capacidad de la célula hospedadora para regular la expresión génica y responder a la infección de manera eficaz.
El coronavirus 2 del síndrome respiratorio agudo grave (SARS-CoV-2) surgió a finales de 2019 y ha demostrado ser un virus altamente virulento que causó la devastadora pandemia mundial de la enfermedad por coronavirus COVID-19.
Trabajos recientes han demostrado que el SARS-CoV-2 altera la regulación epigenética de las células hospedadoras notablemente. Sin embargo, sigue sin estar claro cómo controla el epigenoma de la célula hospedadora y si utiliza la imitación de histonas para hacerlo.
En este trabajo, sus autores examinaron si el SARS-CoV-2 utiliza el mimetismo de histonas para alterar la regulación de la cromatina y la respuesta transcripcional a la infección.
Tras ello, han podido demostrar que la proteína del SARS-CoV-2 codificada por ORF8 funciona como un imitador de histonas de los motivos ARKS en la histona H3 humana, para alterar la regulación epigenética de la célula hospedadora. El gen ORF8 altera la regulación de las modificaciones postraduccionales críticas de las histonas y promueve la compactación de la cromatina. La eliminación de ORF8 o del sitio imitador de histonas, atenúa la capacidad del SARS-CoV-2 para alterar la cromatina de la célula hospedadora, afecta la respuesta transcripcional a la infección y atenúa el número de copias del genoma viral.
Estos hallazgos, demuestran una nueva función de ORF8 y un mecanismo a través del cual el SARS-CoV-2 altera la regulación epigenética de la célula hospedadora. Además, este trabajo proporciona una base molecular para el hallazgo de que la falta de ORF8 en el SARS-CoV-2 está asociada con una menor gravedad del COVID-19.
Métodos de trabajo
Con respecto a los métodos de trabajo o procedimientos que llevaron a cabo los científicos para determinar finalmente cómo el SARS-CoV-2 altera la regulación epigenética de las células hospedadoras, en primer lugar, obtuvieron las células y/o líneas celulares necesarias: las A549 que expresan ACE2 (A549ACE2), las HEK293T, células Vero E6, células epiteliales alveolares inducidas de tipo II (iAT2) derivadas de humanos en formato organoides y células alveolares bidimensionales (2D); así como también obtuvieron el plásmido de expresión de ORF8 y, a partir de este, las construcciones de deleción de ORF8; y una cepa del virus SARS-CoV-2, a partir del cual generaron virus mutantes utilizando el sistema de genética inversa de ADNc.
Posteriormente, cultivaron e infectaron las células con SARS-CoV-2 de tipo salvaje o mutante, a un MOI de 1 PFU por célula (A549ACE2) o 5 PFU por célula (iAT2). Cabe indicar que todas las infecciones y manipulaciones del virus las llevaron a cabo en un laboratorio de nivel de bioseguridad 3 (BSL3) utilizando equipos y protocolos de protección adecuados.
Asimismo, realizaron análisis de cinética de crecimiento y ensayos de placa, recolección de ARN y qRT–PCR y cuantificación del genoma viral. Después de realizar un fraccionamiento celular, determinaron las concentraciones de las muestras y llevaron a cabo un análisis mediante transferencia Western.
Trasfectaron las células HEK293T y las tiñeron para su clasificación y poder generar bibliotecas mediante PCR. Las células transfectadas se aislaron mediante tampón FACS y a las clasificadas se les extrajeron las histonas.
A continuación, les realizaron una secuenciación ATAC (Assay for Transposase-Accessible Chromatin), el cual es un método de secuenciación de alto rendimiento para el análisis de la accesibilidad a la cromatina en todo el genoma. Por su parte, las células A549ACE infectadas se fijaron antes de la recolección para la secuenciación ATAC.
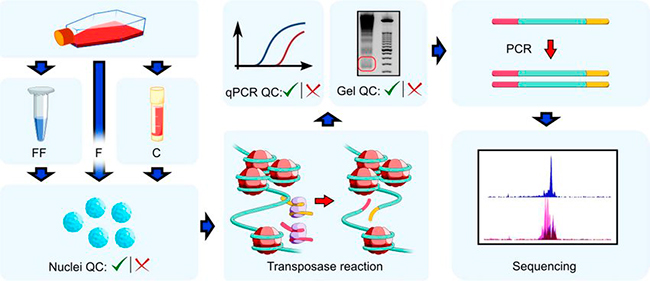
Dos días después de la transfección, llevaron a cabo una inmunoprecipitación de la cromatina acoplada a secuenciación (ChIP-seq) de ORF8, en el que utilizaron perlas magnéticas Strep-Tactin; así como también realizaron ChIP-seq de histona PTM, en el que añadieron un 5% de cromatina de pupas de Camponotus floridanus a todas las muestras. Finalmente, purificaron y extrajeron el ADN para generar bibliotecas mediante PCR.
También extrajeron y purificaron ARN y construyeron un genoma de referencia para la alineación concatenando los genomas humano y SARS-CoV-2 para su análisis.
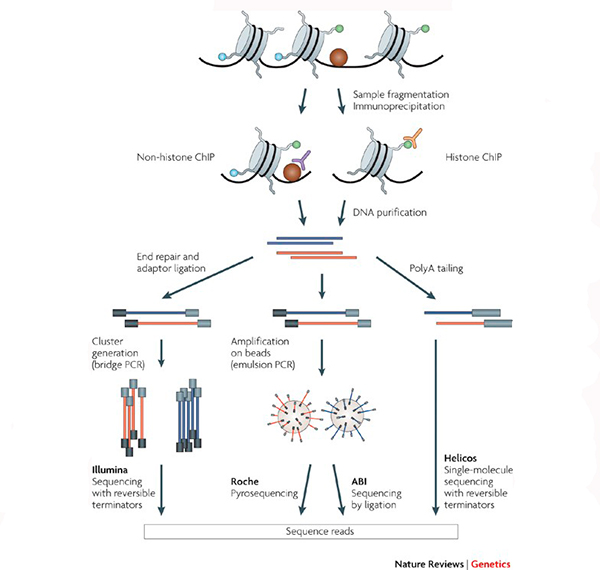
Por otro lado, purificaron por afinidad con Strep Tag la proteína de la cromatina y sus socios de unión, y realizaron una espectrometría de masas para el análisis de ORF8 PTM y los socios de unión. También analizaron los niveles relativos de la construcción ORF8 en fracciones citoplasmáticas frente a las de cromatina mediante transferencia Western, y llevaron a cabo una inmunoprecipitación de la proteína de la cromatina y una inmunoprecipitación inversa de la cromatina.
Por otra parte, realizaron inmunocitoquímica de fluorescencia de las células HEK293T y A549ACE2, donde para la detección de ORF8 marcado con Strep, añadieron Strep-Tactin DY-488 a la solución de anticuerpo secundario, y tiñeron los núcleos con DAPI; así como también hicieron una inmunocitoquímica de fluorescencia de lámina B1, lámina A/C y H3K9me2, utilizando las mismas células HEK293T; cuyas imágenes se obtuvieron mediante un microscopio confocal de barrido láser Leica DM 6000, TCS SP8 vertical con láseres de 405nm, 488nm, 552nm y 638nm, y un sistema confocal de escaneo láser Leica SP8 con un objetivo ×63/1.40-NA HC PL APO CS2 y detectores HyD en modo estándar con una ganancia del 100%. Posteriormente, analizaron las imágenes mediante el software ImageJ.

Para identificar un posible mimetismo de histonas, las secuencias de proteínas del SARS-CoV-2 se alinearon con las secuencias de proteínas de histonas humanas (H2A, H2B, H3.1, H3.2, H4, H2A.X, H2A.Z, macroH2A y H3.3), utilizando la comparación de secuencias múltiples por expectativa logarítmica (MUSCLE) con la configuración predeterminada.
Para el análisis de histonas PTM mediante espectrometría de masas cuantitativa, la purificación de las histonas se validó mediante SDS-PAGE seguida de una tinción con Coomassie que demostró un enriquecimiento suficiente. Se realizó un ensayo BCA para la estimación de proteínas siguiendo las instrucciones del fabricante y se utilizaron 20µg de histona para la derivatización química y la digestión. Las muestras secas se reconstituyeron en ácido trifluoroacético (TFA) y se desalinizaron con la microcolumna de centrifugación C18.
También realizaron una digestión con tripsina y quimotripsina de ORF8 para la identificación de modificaciones de ORF8 y análisis de quimiotripsina por LC-MS/MS y LC-PRM-MS, y una inmunoprecipitación ORF8 versus control para la identificación de los socios de unión; así como realizaron una búsqueda en la base de datos utilizando la secuencia humana SwissProt y la secuencia ORF8 con Proteome Discoverer 2.3 o 2.4, y desarrollaron un método de monitoreo de reacción paralela no programada para identificar o validar posibles dianas peptídicas modificadas y no modificadas de ORF8.
Finalmente, escanearon las muestras y analizaron los datos mediante el software Skyline (MacCoss Lab). También llevaron a cabo una prueba ANOVA y generaron gráficos con correcciones de R. Bonferrroni para comparaciones múltiples.

Resultados del trabajo sobre si el SARS-CoV-2 altera la regulación epigenética
En los siguientes apartados se narran y explican los resultados obtenidos por los científicos en este trabajo.
ORF8 contiene un imitador de la histona H3
Para determinar si el SARS-CoV-2 altera la regulación epigenética mediante un imitador de histona, primero realizaron una comparación bioinformática de todas las proteínas virales del SARS-CoV-2 con todas las proteínas histonas humanas.
Cabe indicar que la mayoría de las proteínas del SARS-CoV-2 son muy similares a las de la cepa de coronavirus que causó el brote principal anterior de SARS-CoV, con la notable excepción de las proteínas codificadas por ORF3b y ORF8, de las cuales ORF8 es la más divergente en el SARS-CoV-2.
Tras ello, detectaron una coincidencia idéntica entre los aminoácidos 50-55 de la proteína codificada por ORF8 y las regiones críticas dentro de la cola N-terminal de la histona H3 (Figura 1a). Además, cabe señalar que ORF8 se alinea con un conjunto secuencial más largo de aminoácidos (seis residuos) que ya fuera previamente descrito y validado con la función de un supuesto mimetismo de histonas en la proteína de la envoltura del SARS-CoV-2. Pero lo más convincente, es que el motivo contiene la secuencia «ARKS», la cual se encuentra en dos sitios distintos en la cola de la histona H3 (como se muestra en la Figura 1a) y está bien establecida como una de las regiones reguladoras más críticas dentro de H3. Ambos sitios ARKS de H3 están modificados con múltiples PTM, que incluyen mono-, di- y trimetilación y acetilación en la lisina 9 de H3 (H3K9me y H3K9ac) y en la lisina 27 de H3 (H3K27me y H3K27ac).
Este tramo de aminoácidos está ausente de la proteína codificada por ORF8 del SARS-CoV anterior, tanto antes como después de que una deleción generara ORF8a y ORF8b25, pero está presente en el SARS-CoV-2 de murciélago y en variantes de interés. ORF8 se expresa en gran medida durante la infección y su transcripción se expresa en niveles más altos que la histona H3 (más del 20% por encima) dentro de las 24h posteriores a la infección.
Finalmente, la caracterización proteómica de los socios de unión de proteínas del SARS-CoV-2, indica que ORF8 se une a la ADN metiltransferasa 1 (DNMT1).
Para determinar si ORF8 funciona como un imitador de histonas, examinaron su localización intracelular. Aunque ORF8 no tiene una secuencia de localización nuclear bien definida, ya que tiene un tamaño de 15 kDa, es lo suficientemente pequeño como para difundirse en el núcleo. Para ello, transfectaron células HEK293T con un constructo que codifica ORF8 marcado con Strep y lo visualizaron con una sonda fluorescente conjugada con Strep-Tactin. Aunque la localización de ORF8 fue variable en apariencia, ORF8 se localizó típicamente en el citoplasma y en la periferia del núcleo cuando se utilizó inmunofluorescencia (Figura 1b), y tanto en el citoplasma como en el núcleo cuando se utilizó fraccionamiento celular.
Dado el patrón de expresión observado, se preguntaron si ORF8 se colocaliza con proteínas laminosas, y descubrieron que ORF8 se colocalizaba con la lamina B1 y la lamina A/C en células transfectadas para expresar ORF8.
A continuación, infectaron una línea celular derivada del epitelio pulmonar A549 que expresaba el receptor ACE2 (A549ACE2) con SARS-CoV-2. Para ello, tiñeron las células con un antisuero específico para ORF8 y confirmaron un patrón de expresión similar en las células infectadas (Figura 1c).
Después, probaron si ORF8 estaba asociado con la cromatina utilizando concentraciones crecientes de sal para examinar la unión de la cromatina. Descubrieron que ORF8 se disocia de la fracción de cromatina en concentraciones de sal similares a aquellas en las que se disocian las proteínas laminosas e histonas (Figura 1d). Por el contrario, ORF8 con una deleción del motivo ARKSAP (ORF8ΔARKSAP) se disoció a concentraciones de sal más bajas y estuvo presente en niveles más bajos en la fracción de cromatina, en comparación con ORF8 con este motivo, lo que indica que el supuesto sitio imitador de histonas afecta a la fuerza de la asociación de ORF8 con la cromatina.
A continuación, realizaron una inmunoprecipitación de cromatina con secuenciación (ChIP–seq) para determinar si ORF8 se asocia con ADN genómico y dónde. Aunque ORF8 no tenía picos claramente definidos, la inmunoprecipitación de ORF8 mostró enriquecimiento sobre la entrada (Figura 1e) y ORF8 se enriqueció dentro de regiones genómicas específicas, particularmente aquellas asociadas con H3K27me3.
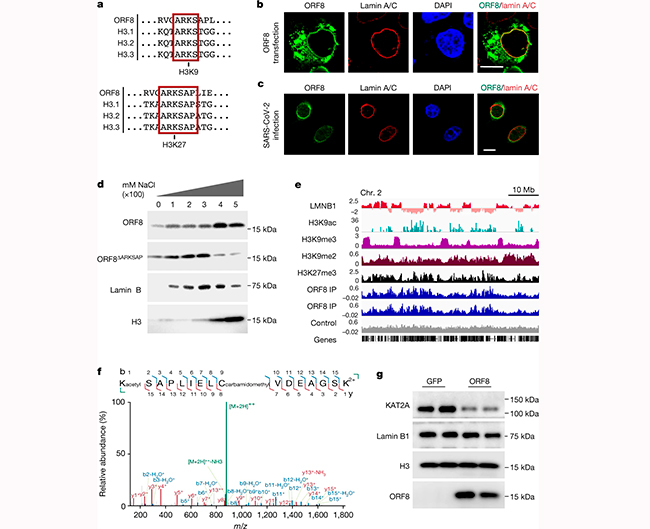
Sobre la base de la localización de ORF8 en la periferia del núcleo y su asociación con la cromatina (observada utilizando enfoques bioquímicos y de secuenciación), probaron además si ORF8 se asociaba con proteínas del complejo laminoso. Descubrieron que ORF8 co-inmunoprecipitaba con la lámina B1, la histona H3 y HP1α, una proteína asociada tanto con proteínas laminosas como con histonas. La co-inmunoprecipitación recíproca para la lámina B1 y la histona H3 confirmó la unión de ORF8.
A continuación, probaron si ORF8 también co-inmunoprecipitaba con las enzimas modificadoras de histonas que se dirigen al motivo ARKS dentro de la histona H3. Descubrieron que ORF8 estaba asociado con la histona acetiltransferasa KAT2A (también conocida como GCN5), que se dirige a H3K9 (Figura 1f). Y aunque tanto ORF8 como ORF8ΔARKSAP se inmunoprecipitaron con un socio de unión citoplasmático previamente establecido, no detectaron la asociación de ORF8ΔARKSAP con proteínas de cromatina, lo que indica que el motivo ARKSAP fortalece la asociación de ORF8 con proteínas de cromatina.
También utilizaron espectrometría de masas para identificar socios de unión adicionales más allá de los encontrados. El lisado de células completas que estaba en gran parte desprovisto de proteínas de cromatina, se utilizó en un enfoque complementario en el que se identificaron principalmente proteínas citoplasmáticas. Sin embargo, se detectó el factor de transcripción SP2 y se confirmó que se unía a ORF8 mediante co-inmunoprecipitación.
Asimismo, sobre la base de la observación de que ORF8 se asocia con KAT2A, utilizaron espectrometría de masas para determinar si el sitio de imitación de histona ORF8 propuesto se modifica de manera similar a las histonas. Utilizando un enfoque de abajo hacia arriba, ORF8 se purificó a partir de células, se redujo, se alquiló y se digirió. La separación con cromatografía líquida fue seguida por espectrometría de masas de monitoreo de reacción paralela (LC-PRM-MS) dirigida a posibles formas no modificadas y modificadas de ORF8 que se encuentran comúnmente para las histonas, incluida la fosforilación de serina y la monometilación, dimetilación, trimetilación y acetilación de lisina.
De estos objetivos, identificaron lisinas no modificadas y acetiladas. El péptido acetilado contenía un cambio de masa de +42 Da y demostró una cobertura casi completa de todos los iones de producto posibles. La espectrometría de masas de alta resolución diferenció el precursor del péptido trimetilado y coincidió con todos los iones de producto dentro de un error de masa de 10 ppm (Figura 1f). Esto demuestra que ORF8 está acetilado en la lisina dentro del sitio de imitación de histona ARKS propuesto, de manera similar a la histona H3. En particular, la presencia de lisina acetilada dentro del motivo ARKSAP sugiere que ORF8 puede existir como un monómero dentro de las células.
Finalmente, dado que ORF8 promueve la degradación lisosomal de otro socio de unión, examinaron si ORF8 afectaba de manera similar a las proteínas asociadas a la cromatina. La expresión de ORF8 resultó en una marcada disminución en la abundancia de KAT2A (Figura 1g), mientras que los niveles de proteínas de la lámina nuclear y heterocromatina asociada a la lámina no cambiaron. Estos hallazgos, sugieren que ORF8 no solo se asocia con proteínas como las acetiltransferasas, sino que probablemente también es modificado por ellas de manera similar a la histona H3 e induce su degradación.
En conjunto, estos hallazgos demuestran que ORF8 está bien posicionado para actuar como un imitador de histonas sobre la base de su asociación con la cromatina y las enzimas modificadoras de la cromatina y su capacidad para agotar la acetiltransferasa de histonas KAT2A.
ORF8 altera la regulación de la cromatina
A continuación, examinaron si la expresión de ORF8 altera las PTM de histonas utilizando un enfoque de espectrometría de masas imparcial. Para ello, las células HEK293T se transfectaron con un plásmido de control que codifica GFP o con un plásmido que codifica ORF8 con una etiqueta Strep. Las células transfectadas identificadas por fluorescencia de GFP o por una sonda fluorescente conjugada con Strep-Tactin, se aislaron utilizando la clasificación celular activada por fluorescencia (FACS). Las histonas se purificaron mediante extracción ácida y se realizó una espectrometría de masas imparcial de abajo a arriba para cuantificar todas las PTM de histonas detectadas.
Las modificaciones de histonas asociadas con la represión transcripcional aumentaron, mientras que numerosas modificaciones de histonas asociadas con la expresión génica activa se agotaron en las células que expresaban ORF8 (Figura 2a). En particular, las modificaciones dentro de los motivos ARKS de H3 se vieron altamente alteradas. Por ejemplo, los péptidos que contienen H3K9 y H3K27 metilados, que están asociados con la represión transcripcional, mostraron una abundancia aumentada de manera robusta en respuesta a la expresión de ORF8. Por el contrario, el péptido que contiene tanto H3K9ac como H3K14ac, los cuales tienen ambos un vínculo bien establecido con la expresión génica activa, mostró una abundancia disminuida en respuesta a la expresión de ORF8. Estos datos respaldan un papel para ORF8 como un supuesto imitador de histonas y demuestran que es capaz de alterar la regulación de PTM de histonas en numerosos sitios críticos dentro de estas.
Para confirmar los hallazgos de la espectrometría de masas, utilizaron imágenes de inmunofluorescencia para medir H3K9 y H3K27 metilados y acetilados. Descubrieron que las células que expresaban ORF8 exhibieron un aumento de H3K9me3 y H3K27me3, y una disminución de la tinción de H3K9ac, en comparación con las transfectadas con el plásmido de control (Figuras 2b–g). Y aunque ORF8ΔARKSAP se expresó en niveles similares a ORF8, no aumentó H3K9me3 ni H3K27me3 y tuvo un efecto intermedio no significativo en H3K9ac.
A continuación, examinaron una mutación adquirida en ORF8 que se encuentra comúnmente en las cepas de SARS-CoV-2 que codifica una sustitución S84L (ORF8S84L). Es poco probable que este sitio afecte la estabilidad de la proteína y se encuentra fuera de la región imitadora de histonas, por lo que no se espera que la sustitución afecte la capacidad de ORF8 para regular las PTM de histonas. Así, la expresión de ORF8S84L también aumentó los niveles de H3K9me3 y H3K27me3, mientras que disminuyó H3K9ac, lo que indica que, como se predijo, esta variante común no altera la función imitadora de histonas de ORF8. De manera similar, una deleción de seis residuos en otra región no estructurada de ORF8 con una composición de aminoácidos similar pero una secuencia diferente (AGSKSP), tampoco afectó la capacidad de ORF8 para alterar la regulación de histonas.
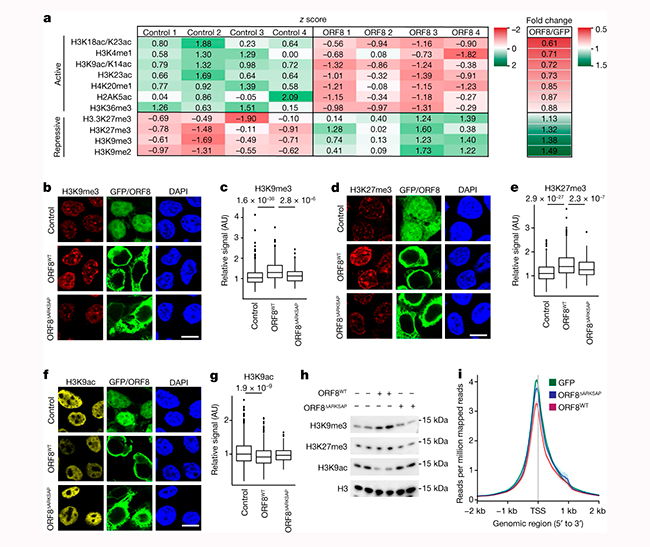
Posteriormente, buscaron confirmar estos hallazgos utilizando métodos independientes. Para asegurar niveles iguales de expresión de ORF8 y ORF8ΔARKSAP, aislaron las células transfectadas mediante tampón FACS. Luego, aislaron las histonas mediante extracción ácida y confirmaron que ORF8 aumentó H3K9me3 y H3K27me3 y eliminó H3K9ac de una manera dependiente de ARKSAP, mediante análisis de transferencia Western (Figura 2h).
Finalmente, el ensayo de cromatina accesible a la transposasa con secuenciación de alto rendimiento (ATAC-seq), demostró que ORF8, pero no ORF8ΔARKSAP, disminuyó la accesibilidad de la cromatina. Los cambios tanto en H3K9ac como en la accesibilidad de la cromatina, fueron en gran medida globales, pero fueron particularmente evidentes para los genes con expresión intermedia a alta.
Para determinar cómo estas alteraciones de la cromatina afectaban la expresión génica, utilizaron la secuenciación de ARN (RNA-seq) para definir genes expresados de manera diferencial en células transfectadas. Si bien ORF8 y ORF8ΔARKSAP compartían un subconjunto de genes expresados de manera diferencial, la presencia del motivo imitador de histonas resultó en cambios de expresión génica menos dinámicos. También se expresaron de manera diferencial grupos de genes distintos entre ORF8 y ORF8ΔARKSAP, y ORF8 disminuyó la expresión génica en relación con ORF8ΔARKSAP, en particular los genes altamente expresados. Cabe señalar que los genes que se regularon a la baja en respuesta a la expresión de ORF8 en relación con ORF8ΔARKSAP, también tenían niveles basales más altos de H3K9ac y mayor accesibilidad que los genes que se regularon al alza, lo que sugiere que pueden ser más sensibles al agotamiento de H3K9ac.
En conjunto, estos resultados respaldan un modelo en el que ORF8 tiene múltiples funciones y activa varias vías de expresión génica, en particular en ausencia del motivo ARKSAP. Sin embargo, la presencia del motivo ARKSAP amortigua la respuesta transcripcional de la célula hospedadora y disminuye la expresión de genes con alta accesibilidad y H3K9ac. Además, estos datos definen un papel para ORF8 en la alteración de las PTM de histonas de la célula hospedadora a través de un nuevo caso de mimetismo de histonas de los motivos ARKS en la histona H3.

El SARS-CoV-2 altera la regulación de la cromatina
Tras demostrar que ORF8 por sí solo es suficiente para alterar la regulación de la cromatina, examinaron el efecto de ORF8 en la regulación de la PTM de la histona en el contexto de la infección viral. Para ello, generaron un virus SARS-CoV-2 mutante recombinante con una deleción de ORF8 (SARS-CoV-2ΔORF8) utilizando un sistema de genética inversa de ADNc. Infectaron células A549ACE2 con SARS-CoV-2 o SARS-CoV-2ΔORF8, y compararon los niveles de los genomas virales y la producción de virus infecciosos en presencia y ausencia de ORF8. También utilizaron ChIP-seq con normalización de picos (ChIP-RX) para la detección de cambios globales en los PTM de histonas.
No detectaron diferencias en el número de copias del genoma ni en el título viral a las 24h, y solo observaron diferencias sutiles a las 48h, lo que permitió una comparación directa de estos dos virus en estos puntos temporales tempranos después de la infección.
Descubrieron que la infección por SARS-CoV-2 resultó en aumentos robustos de H3K9me3 y H3K27me3, en comparación con las células infectadas simuladas, lo que refleja los efectos de la expresión de ORF8. Sin embargo, la eliminación de ORF8 atenuó sustancialmente este efecto, lo que indica que el efecto del SARS-CoV-2 sobre las modificaciones represivas de las histonas se debe en parte a la expresión de ORF8.
De manera similar, ATAC-seq demostró que la infección con SARS-CoV-2 de tipo salvaje resultó en una condensación sustancial de la cromatina, mientras que la infección con SARS-CoV-2ΔORF8 resultó en un fenotipo intermedio.
Finalmente, ChIP-RX indicó que la infección por SARS-CoV-2 resultó en una disminución de H3K9ac, y este efecto se atenuó nuevamente en células infectadas con SARS-CoV-2ΔORF8 (Figuras 3d, e).
Todos estos datos, demuestran que ORF8 contribuye a los efectos de la infección por SARS-CoV-2 en la accesibilidad de la cromatina y las modificaciones de las histonas en las células hospedadoras.
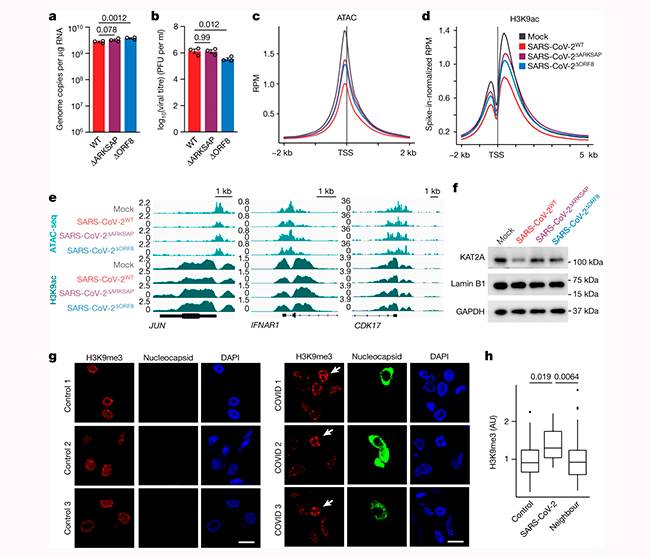
Debido a que es probable que ORF8 tenga múltiples efectos sobre la función celular, también buscaron determinar si estos efectos se debían específicamente al motivo imitador de histonas. Para ello, generaron una forma mutante del SARS-CoV-2 con una eliminación de solo el motivo ARKSAP (SARS-CoV-2ΔARKSAP). En las células A549ACE2, el SARS-CoV-2ΔARKSAP se replicó de manera similar al virus de tipo salvaje (Figuras 3a, b) pero alivió sustancialmente el efecto de la infección en la accesibilidad de la cromatina y H3K9ac, coincidiendo con los efectos de la eliminación de ORF8 (Figuras 3c–e).
Dados los fuertes efectos del SARS-CoV-2 en H3K9ac y la capacidad de ORF8 para agotar KAT2A, también examinaron el efecto de la infección en los niveles de KAT2A. La infección por SARS-CoV-2 de tipo salvaje redujo la expresión de KAT2A, mientras que la infección con SARS-CoV-2ΔORF8 o SARS-CoV-2ΔARKSAP no lo hizo (Figura 3f). Estos datos indican que ORF8, y específicamente el motivo ARKSAP dentro de ORF8, contribuye a los efectos del SARS-CoV-2 en el epigenoma de la célula hospedadora.
Para asegurar que las diferencias observadas en la regulación de la cromatina de la célula hospedadora después de la infección por SARS-CoV-2 y SARS-CoV-2ΔORF8 no se debieran a ninguna diferencia sutil en las tasas de infección entre los virus, buscaron confirmar aún más estos hallazgos utilizando un enfoque independiente del número de células infectadas. Por ello, utilizaron inmunocitoquímica para teñir las modificaciones de histonas de interés, utilizando tinción para ARN bicatenario (dsRNA) para identificar y examinar específicamente las células infectadas.
A las 24h después de la infección, las células infectadas con SARS-CoV-2 habían aumentado H3K9me3 y H3K27me3 y disminuido H3K9ac, en comparación con las células infectadas simuladas o las células vecinas no infectadas. Como se observó en los datos de ChIP-seq, este efecto se perdió en gran medida con la eliminación de ORF8.
Para determinar si también se producen efectos similares en el contexto de una población de pacientes, obtuvieron muestras de tejido pulmonar post mortem de tres pacientes con enfermedad por coronavirus 2019 (COVID-19) y controles emparejados. Tiñeron el tejido para H3K9me3, así como para la proteína de la nucleocápside del SARS-CoV-2, para identificar las células infectadas. Descubrieron que, en todas las muestras de pacientes, las células infectadas mostraron un aumento de la tinción de H3K9me3, en comparación con las células vecinas dentro del mismo tejido, así como en comparación con el tejido de control (Figuras 3g, h).
Si bien la disponibilidad de la muestra limita las conclusiones que se pueden extraer de este ensayo, este hallazgo indica que las PTM de histonas también se alteran en pacientes con enfermedad grave por COVID-19. En resumen, descubrieron que los efectos de la infección por SARS-CoV-2 sobre las PTM de histonas y la compactación de la cromatina, requieren la expresión de ORF8 y reflejan los efectos dependientes de ARKSAP de ORF8.
Efectos del SARS-CoV-2 en la transcripción
A continuación, examinaron cómo los cambios en las PTM de histonas detectados a través de ChIP-seq se relacionaban con la expresión génica mediante RNA-seq. Todos los virus contenían cantidades similares de lecturas, y la única diferencia en la expresión de la transcripción del SARS-CoV-2 era para ORF8 en SARS-CoV-2ΔORF8. Sin embargo, en el virus de tipo salvaje, la transcripción de ORF8 se expresó en gran medida y era más abundante que las transcripciones que codifican la histona H3.
Curiosamente, al principio de la infección, los tres virus analizados alteraron cada uno un conjunto distinto de genes, lo que indica que la presencia del motivo imitador de la histona cambia la respuesta transcripcional a la infección (Figura 4a–c). A las 48h después de la infección, los tres virus constituyeron la gran mayoría de las lecturas mapeadas y dieron como resultado cambios robustos en la expresión génica, en comparación con las células infectadas de forma simulada. Los grupos funcionales de genes más inducidos por la infección también diferían entre los tres virus, lo que indica respuestas distintas de las células hospedadoras en puntos temporales tempranos (Figura 4d). Esto es notable dado que el SARS-CoV-2 de tipo salvaje y el SARS-CoV-2ΔARKSAP tenían números de copias y tasas de replicación casi idénticos en células A549ACE2 (Figura 3a, b), y por lo tanto, es poco probable que las diferentes respuestas transcripcionales se deban a diferencias en el número de células infectadas o la carga viral dentro de las células infectadas. Además, la comparación directa de SARS-CoV-2ΔORF8 y SARS-CoV-2ΔARKSAP también mostró cambios distintos en la expresión génica y enriquecimiento del grupo funcional, lo que indica nuevamente que ORF8 probablemente tiene múltiples funciones más allá de las mediadas por el dominio ARKSAP. Adicionalmente, los cambios en la expresión génica en respuesta a la infección se correlacionaron con cambios en H3K9ac.
Por tanto, cabe destacar que estos datos respaldan aún más los hallazgos recientes que indican que el SARS-CoV-2 produce una respuesta transcripcional temprana limitada y demuestran que el dominio ORF8 ARKSAP está vinculado a cambios en la expresión génica.
Dados los sólidos efectos de la eliminación de ORF8 en la regulación de la cromatina de la célula hospedadora y la respuesta transcripcional a la infección, buscaron probar si ORF8 medía la replicación del SARS-CoV-2 utilizando un tipo de célula fisiológicamente relevante. Para ello, infectaron células pulmonares alveolares pulmonares de tipo II (iAT2) derivadas de células madre pluripotentes humanas inducidas con SARS-CoV-2, SARS-CoV-2ΔORF8 o SARS-CoV-2ΔARKSAP (multiplicidad de infección (MOI) = 1).
Tras esto, observaron que ambos virus mutantes habían disminuido el número de copias del genoma a las 48h después de la infección en la mayoría de las réplicas (Figura 4e), lo que sugiere que ORF8, y específicamente el dominio ARKSAP, afecta la replicación del genoma del SARS-CoV-2 en una célula hospedadora. Sin embargo, los títulos virales medidos a través de ensayos de placa, demostraron que SARS-CoV-2ΔORF8 generó menos partículas infecciosas que el SARS-CoV-2 de tipo salvaje, mientras que SARS-CoV-2ΔARKSAP parecía similar al virus de tipo salvaje y en algunos casos incluso mostró una mayor formación de placa (Figura 4f).
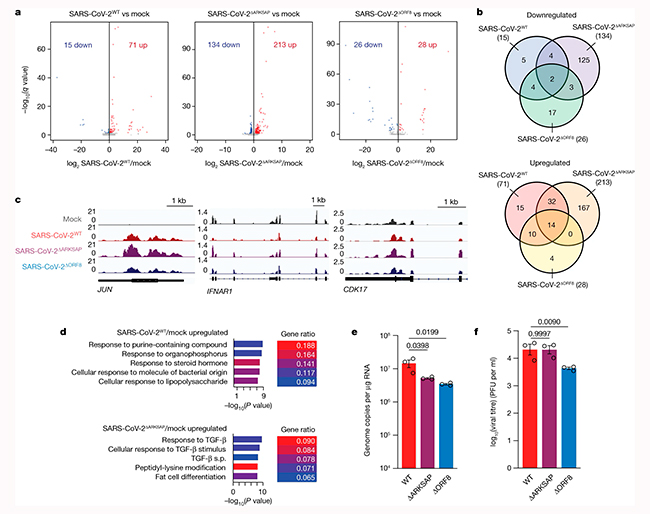
En consonancia con el trabajo previo que indicaba que ORF8 afecta las vías de estrés del retículo endoplásmico, esto sugiere que ORF8 probablemente tiene una función independiente de ARKSAP que puede promover la formación de partículas virales.
En conjunto, este trabajo presenta un vínculo entre una proteína específica del SARS-CoV-2 y las alteraciones epigenéticas que ocurren en respuesta a la infección y proporciona una explicación mecanicista de la creciente evidencia de que las alteraciones epigenéticas contribuyen a la gravedad de la COVID-19.
Discusión
Este trabajo identifica un nuevo caso de mimetismo de histonas durante la infección por SARS-CoV-2 y define un mecanismo a través del cual el SARS-CoV-2 actúa para interrumpir la regulación de la cromatina de la célula hospedadora.
Se descubrió que la proteína codificada por el gen ORF8 del SARS-CoV-2 contiene un motivo ARKS y que la expresión de ORF8 interrumpe la regulación de las PTM de las histonas.
ORF8 está asociado con proteínas asociadas a la cromatina, histonas y la lámina nuclear, y está acetilado dentro del motivo mimético de histonas de manera similar a las histonas. La expresión de ORF8 interrumpe múltiples PTM de histonas críticas y promueve la compactación de la cromatina, mientras que el ORF8 que carece del motivo mimético de histonas no lo hace.
Además, la infección por SARS-CoV-2 en líneas celulares humanas y tejido pulmonar de pacientes post mortem, causa alteraciones globales a la cromatina similares que actúan, en parte, a través del mimetismo de histonas. Asimismo, la eliminación del gen ORF8 o la secuencia que codifica el mimético de histonas afecta la respuesta transcripcional de la célula hospedadora a la infección por SARS-CoV-2. Por último, la pérdida de ORF8 disminuye la replicación del SARS-CoV-2 en células pulmonares iAT2 derivadas de células madre pluripotentes inducidas humanas, mientras que la pérdida del motivo imitador de histonas afecta específicamente al número de copias del genoma viral.

Cabe destacar que el papel de ORF8 en la alteración de la cromatina al principio de la infección, no es incompatible con otros papeles propuestos para ORF8 en otros compartimentos celulares o en etapas posteriores de la infección, así como no excluye otros mecanismos propuestos de alteración transcripcional en respuesta al SARS-CoV-2. De hecho, se apunta hacia un modelo en el que ORF8 tiene múltiples funciones, incluida la de actuar como un motivo imitador de histonas.
Asimismo, los hallazgos también proporcionan un mecanismo epigenético subyacente al papel de ORF8 en la promoción de la virulencia del SARS-CoV-2 dentro de la población de pacientes.
Finalmente, el trabajo tiene implicaciones críticas para comprender las cepas virales emergentes que presentan deleciones y mutaciones en el gen ORF850 y la patogénesis de COVID-19 en pacientes.
Fuente de este artículo
Este artículo fue publicado originalmente en la revista Nature bajo la referencia: Kee, J., Thudium, S., Renner, D.M. et al. SARS-CoV-2 disrupts host epigenetic regulation via histone mimicry. Nature 610, 381–388 (2022). https://doi.org/10.1038/s41586-022-05282-z; estando disponible con acceso libre en la propia página de Nature.


