Los sistemas antivirales dirigidos por ARN pequeño están activos en mamíferos y en otros organismos. En este artículo, se resume el conocimiento actual de las funciones antivirales de cada uno de estos pequeños tipos de ARN, así como se analiza el potencial para el desarrollo de terapias y profilaxis antivirales dirigidas por ARN pequeño no codificante.
Presentación
“Mammalian antiviral systems directed by small RNA” (“Sistemas antivirales en mamíferos dirigidos por ARN pequeños”) es un artículo científico elaborado por Takahashi, T., Heaton, S. M. y Parrish, N. F., publicado en Diciembre de 2021 en la revista PLOS Pathogens, en el que se resume el conocimiento actual de las funciones antivirales de cada uno de los tipos de ARN de sistemas antivirales dirigidos por ARN pequeño, así como se analiza el potencial para el desarrollo de terapias y profilaxis antivirales dirigidas por ARN pequeño no codificante.
Los virus son la entidad más abundante y genéticamente diversa en nuestra biosfera y existen dondequiera que haya vida. Se caracterizan por depender de productos proteínicos codificados por el genoma del hospedador y otros materiales (p. ej., la membrana plasmática) para su replicación, mientras que la replicación celular y del organismo del hospedador es, en principio, independiente del material genético de los virus. La replicación de algunos virus está asociada con la muerte celular y del organismo.
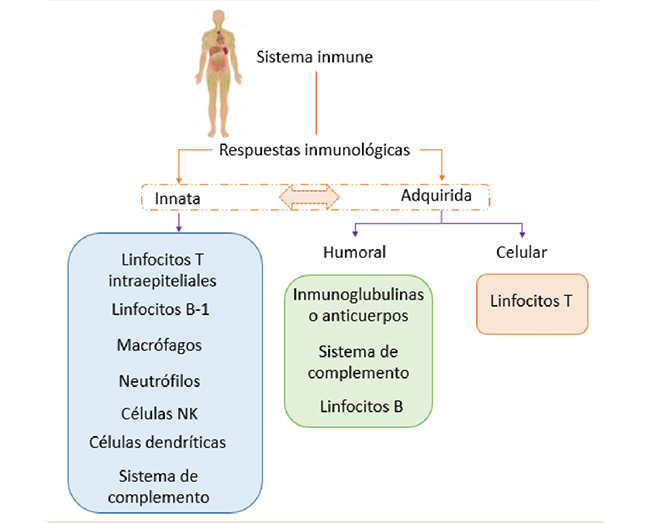
En los mamíferos, los sistemas inmunitarios mejor estudiados están dirigidos por proteínas que reconocen específicamente a los virus. Los sistemas inmunitarios capaces de distinguir lo “propio” de lo “ajeno” y eliminar lo “ajeno” se han establecido y refinado a través de interacciones antagónicas entre los virus y sus hospedadores durante largos períodos de tiempo. Los organismos se han adaptado y codifican varios sistemas inmunitarios para limitar la replicación del virus. Los componentes de los sistemas inmunitarios de los mamíferos suelen clasificarse como pertenecientes a la respuesta innata o adaptativa, en función de características funcionales y evolutivas generalizables.
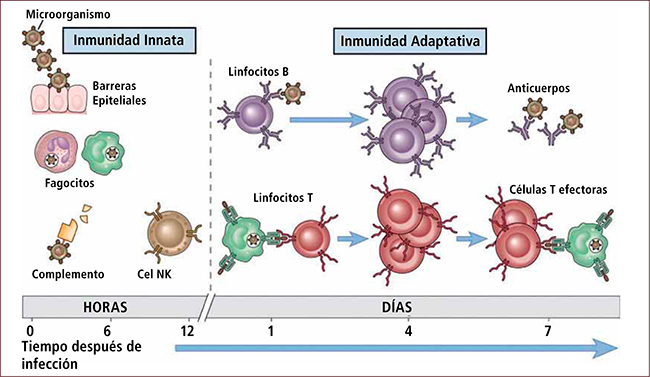
Cuando un virus infecta células humanas, primero se activa la inmunidad innata, seguida de la inmunidad adaptativa. La innata se activa mediante características comunes a los patógenos invasores, denominadas patrones moleculares asociados a patógenos (PAMP), los cuales son reconocidos por proteínas sensoras como los receptores tipo Toll (TLR) y los receptores tipo RIG-I (RLR). Tras la activación, estos receptores inducen la producción de varias citocinas antivirales, como el interferón tipo I (IFN), que estimulan la expresión de genes antivirales que reprimen la replicación viral y reclutan o activan células especializadas, incluidos los fagocitos y las células presentadoras de antígenos. Los linfocitos inmunitarios adaptativos generan receptores muy diversos a través de la recombinación genética y luego proliferan selectivamente en función de la especificidad de su receptor. Esto permite una alta especificidad, así como propiedades anamnésicas que dependen de células de «memoria» de larga vida.
La inmunidad adaptativa convencional de los mamíferos se dirige a los antígenos proteicos presentados y se caracteriza por una novedad evolutiva relativa; los genes críticos se conservan solo entre los vertebrados con mandíbulas, y la mutación por pérdida de función de algunos genes inmunes se ha asociado con una mayor susceptibilidad a la infección viral fatal antes de la madurez reproductiva, lo que argumenta firmemente que la actividad antiviral se encuentra entre sus funciones evolucionadas.
Por su parte, los sistemas inmunitarios antivirales dirigidos por ARN pequeño suprimen la invasión de células por material genético ajeno a sí mismo mediante el apareamiento de bases complementarias con secuencias diana. Estos sistemas dependientes del silenciamiento del ARN operan en diversos organismos. Así pues, en los mamíferos, existe evidencia sólida de que los microARN (miARN) regulan genes endógenos importantes para la inmunidad antiviral, y hay evidencia emergente de que los ácidos nucleicos derivados de virus pueden ser atacados directamente por ARN interferentes pequeños (siARN), ARN que interactúa con PIWI (piARN) y ARN de transferencia (ARNt), para protección en algunos contextos. En otros organismos, incluidas las bacterias, los sistemas inmunitarios dirigidos por ARN pequeño también son bien conocidos, los ARN guía derivados de espaciadores en los sistemas inmunitarios CRISPR/Cas son un ejemplo destacado.
Algunas respuestas dirigidas por ARN pequeños se amplifican de una manera que recuerda la expansión de clones de linfocitos específicos de virus en la inmunidad adaptativa, sin embargo, los mecanismos inmunitarios dirigidos por ARN pequeño descritos desafían la categorización como innatos o adaptativos; por ejemplo, algunos pueden responder inmediatamente después de la infección viral, pero también tienen una alta especificidad mediada por el apareamiento de bases complementarias con sus secuencias objetivo, tal y como hemos señalado.
En lugar de haber «cambiado» estos sistemas por los dirigidos por proteínas más avanzados, cada vez hay más pruebas que respaldan la perspectiva de que los sistemas antivirales dirigidos por ARN pequeño siguen activos en la inmunidad antiviral de los mamíferos en algunos contextos.
En este artículo, se resume el conocimiento actual de las funciones antivirales de cada uno de estos pequeños tipos de ARN agrupados sobre la base de su biogénesis, considerando su superposición conceptual y mecanicista con la inmunidad innata y adaptativa guiada por proteínas, así como se analiza el potencial para el desarrollo de terapias y profilaxis antivirales dirigidas por ARN no codificante pequeño.
Eliminación de ARN viral mediante ARN de interferencia dirigido por siRNA
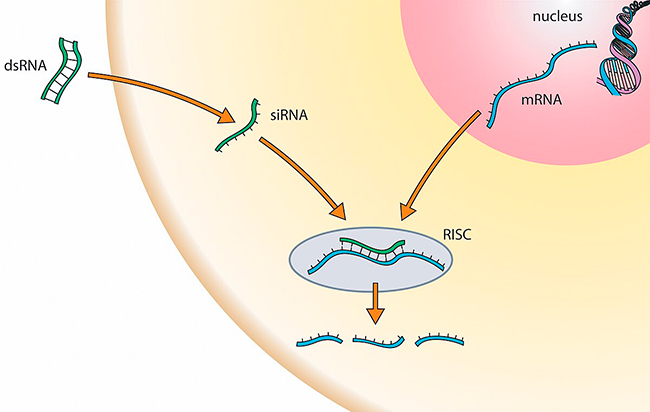
El ejemplo mejor estudiado de inmunidad o de sistemas antivirales dirigidos por ARN pequeño es el guiado por siRNA. Cuando los virus de ARN o ADN infectan células, se pueden generar ARN bicatenarios de origen viral (ARNbc) como intermediarios de replicación o como regiones estructuradas de material genético viral. Estos ARNbc pueden ser escindidos por la endorribonucleasa Dicer, en siRNA, que tiene una longitud característica de 21 a 24 nucleótidos (nt) con un saliente 3′ de 2 nt.
Una de las hebras resultantes de la escisión, la hebra guía, se carga en una proteína argonauta (AGO), también una endonucleasa, y se aparea con el ARN viral complementario. Los AGO son componentes centrales del complejo de silenciamiento inducido por ARN (RISC) y algunos desencadenan la escisión del ARN viral, lo que da como resultado la represión de la replicación viral. Aquí, se refieren a este sistema como ARNi antiviral, y funciona como un potente sistema de defensa en plantas e invertebrados.
Sin embargo, es controvertido si el ARNi antiviral funciona como un aspecto significativo de las defensas de los mamíferos contra los virus. Revisiones recientes han catalogado la evidencia a favor de esta actividad y, desde 2020, experimentos con ratones han demostrado que AGO4 está involucrado en la defensa contra el virus de la influenza y AGO2 contra el virus Nodamura (NoV).
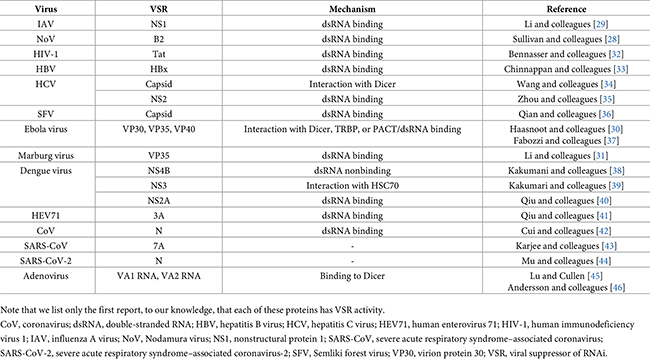
Los mecanismos propuestos para limitar la eficacia de los ARNi antivirales en mamíferos son potencialmente relevantes para la actividad antiviral de otros ARN pequeños. Según se informa, dos tipos de mecanismos, los habilitados por los supresores virales de ARNi (VSR) y la señalización de IFN, limitan la efectividad de los ARNi en la inmunidad de los mamíferos.
La eliminación o mutación de VSR, como la proteína B2 de NoV o la proteína no estructural 1 (NS1) del virus de la influenza A (IAV), conduce a la producción de siRNA derivados de virus (vsiRNA) en células somáticas humanas o de ratón, mientras que la infección por los virus intactos no lo hace (Tabla 2). Varias proteínas, como la proteína 35 del virión (VP35) del virus del Ébola o Marburgo, pueden prevenir la acumulación de vsiRNA en células infectadas por virus que carecen de su VSR nativo. Además, la eliminación de estos VSR puede reprimir la replicación viral, y la represión se recupera por la deficiencia de Dicer o AGO2. Asimismo, los péptidos inhibidores VSR pueden inducir la producción de vsiRNA, lo que sugiere que los VSR aumentan la replicación viral al inhibir el procesamiento de vsiRNA.
Sin embargo, el Dicer humano también regula la maduración de miRNA endógenos, por lo que se puede suponer que la deficiencia de Dicer también afecta la regulación génica postranscripcional por miRNA endógenos, mientras que la deficiencia de AGO2 tiene poco efecto en la población general de miRNA. En otras palabras, incluso en entornos en los que el Dicer humano tiene un efecto antiviral, es difícil interpretar si este se deriva de la escisión del ARN viral por vsiRNA o de la regulación de la expresión génica antiviral por miRNA endógenos.
Se ha demostrado que los VSR actúan en varios pasos en las vías de ARNi antiviral, incluido el secuestro de ARNbc y la inhibición de los factores de silenciamiento del ARN mediante la interacción ARN/proteína o proteína/proteína.
Por otro lado, como se ha indicado, el antagonismo entre las vías de IFN y ARNi también puede limitar la eficacia de esta última. La eliminación de MAVS (también conocida como IPS-1), una proteína adaptadora esencial en la señalización de IFN, permite el ARNi inducido por dsRNA, el cual se pierde nuevamente tras la pérdida o disminución de AGO2, lo que indica que la señalización de IFN enmascara el ARNi inducido por dsRNA dependiente de AGO2 en células somáticas de mamíferos.
Algunos genes estimulados por IFN (ISG) codificados en genomas de mamíferos podrían funcionar como supresores endógenos de ARNi, ya que el tratamiento con IFN reprime el procesamiento de ARN pequeño por Dicer en células no infectadas. Se ha demostrado que al menos un ISG, LGP2, reprime la actividad de Dicer, pero el IFN estimula cientos de ISG, por lo que es posible que otros ISG también estén involucrados en la supresión de ARNi.
Si bien los efectos provocados por el IFN pueden bloquear la producción de vsiRNA en algunos entornos, el antagonismo por parte del IFN puede no ser la única explicación de la actividad restringida del ARNi antiviral en mamíferos. Por ejemplo, la eliminación de RIG-I o MDA5 anuló la producción de IFN, pero no descubrió la producción de vsiRNA durante la infección de células HeLa por virus de ARN de una cepa de vacuna del virus de la fiebre amarilla 17D y coxsackievirus B3 (CBV3), lo que sostiene que la actividad supresora de los ISG no puede evocarse como la razón por la que los vsiRNA no se producen en esas condiciones.
Por otro lado, mientras que en general las células diferenciadas producen IFN tras la infección viral o el tratamiento con poli(I:C), algunas células madre pluripotentes o multipotentes no lo hacen y muestran una respuesta atenuada al IFN exógeno. Las células madre pluripotentes pueden procesar el dsRNA viral en vsiRNA durante la infección por EMCV o NoV, y la producción de vsiRNA es inhibida por la proteína B2 de NoV. Las células madre embrionarias de ratón (mESC) tienen una respuesta de IFN atenuada, pero muestran ARNi inducido por dsRNA, mientras que esta actividad no se detecta después de la diferenciación. También las células progenitoras neuronales de ratón infectadas con el virus Zika (ZIKV) o SINV mostraron producción de vsiRNAs.
El reciente descubrimiento de isoformas alternativas de Dicer con actividad antiviral en células madre humanas y de ratón extiende este concepto. Así, es posible que algunas células madre tengan actividad antiviral de ARNi, pero otras no. Además, al interpretar el efecto inhibidor de IFN o ISG para ARNi antiviral, es importante considerar si la represión traduccional global ha sido inducida por la proteína quinasa R (PKR) en las condiciones utilizadas. PKR es activada por dsRNA viral e induce represión traduccional global, y la activación de PKR puede ocurrir aguas arriba y potencialmente de forma independiente de la señalización de IFN. Se ha informado que la PKR se activa constitutivamente en las mESC deficientes en Dicer y que la producción de IFN se eleva en las mismas, lo que sugiere que la replicación viral diferencial en las mESC deficientes en Dicer resulta de la combinación de la eliminación del ARN viral por el ARNi antiviral y la represión traduccional inducida por PKR de las proteínas virales utilizadas en la replicación viral.
En el contexto de la activación de PKR, la producción de IFN puede no corresponder necesariamente a una alta expresión de proteínas de ISG. Curiosamente, también se ha informado que varios VSR actúan como represores de la producción de IFN y represores de la activación de PKR al interferir con su actividad de unión al dsRNA.
En resumen, la acumulación de dsRNA tiene varias consecuencias que limitan la permisividad celular a la replicación del virus. Sin embargo, las interacciones entre las vías sensibles al dsRNA impiden realizar afirmaciones simples sobre la relevancia fisiológica de los vsiRNA de mamíferos, especialmente aquellas que caracterizarían a los vsiRNA como diferentes de las vías inducidas simultáneamente y reconocidas desde hace tiempo como antivirales, como el IFN.
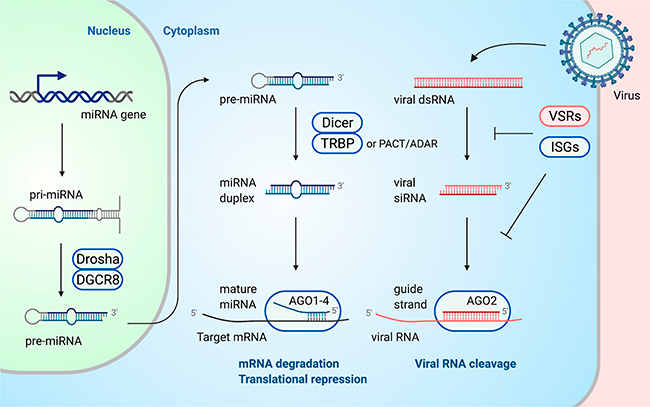
Regulación génica postranscripcional mediante silenciamiento de ARN dirigido por miRNA
Los miRNA dirigen la regulación génica postranscripcional y, al hacerlo, a menudo, regulan las respuestas antivirales de una manera consistente con que esta sea una de sus funciones evolucionadas. Los miRNA exhiben patrones de expresión específicos de cada tejido, lo que sugiere que los miRNA están involucrados en las complejas redes reguladoras de genes características de cada tejido. La maquinaria de biogénesis de miRNA a veces está directamente involucrada en la defensa antiviral; Dicer regula tanto la producción de vsiRNA como de miRNA en células infectadas por virus, y se informa que Drosha es capaz de escindir estructuras similares a pri-miRNA en ARN virales. Los miRNA no inducen la escisión del ARN incluso si se incorporan a AGO2, sino que regulan selectivamente los ARN desestabilizándolos o reprimiendo su traducción. Algunos miRNA se dirigen a los ARNm endógenos de una manera que promueve respuestas antivirales, mientras que otros se dirigen directamente a los ARN virales (ver Tabla 3 de ejemplos de inmunidad antiviral dirigida por miRNA endógenos).
En cuanto al siRNA, también se ha informado de una regulación mutua con el sistema IFN para el miRNA. La estimulación con IFN conduce a una regulación distinta de los grupos de miRNA, con algunos aumentos y otras disminuciones. Y además de cambiar la abundancia, la modificación epitranscripcional de los miRNA puede influir en sus actividades antivirales.
Tanto la regulación global como la de poblaciones específicas de miRNA, pueden mejorar la eliminación del virus o vincular los sistemas inmunológicos innato y adaptativo. La disminución global de los miRNA induce la producción de citocinas proinflamatorias. La disminución específica en el grupo de miRNA unidos por TRBP regula positivamente los genes reguladores de la apoptosis y da como resultado un aumento de la apoptosis de las células infectadas por el virus.
Los miRNA también pueden liberarse de las células infectadas y transportarse en vesículas extracelulares, lo que sugiere la posibilidad de que los miRNA funcionen como citocinas para transmitir señales y modificar la expresión génica de otras células. La dinámica de los miRNA influye tanto en las redes intracelulares como intercelulares relevantes para los estados antivirales. Al igual que los niveles de citocinas o los títulos de anticuerpos, el seguimiento de los miRNA empaquetados en vesículas extracelulares se ha propuesto como un biomarcador para la predicción de la eficacia antiviral; por ejemplo, los niveles séricos de miR-122 en pacientes infectados con VHC reflejan el éxito de la terapia con IFN/ribavirina. Por último, algunos virus codifican miRNA, entendiéndose que estos contrarrestan la inmunidad antiviral y promueven la replicación del virus.
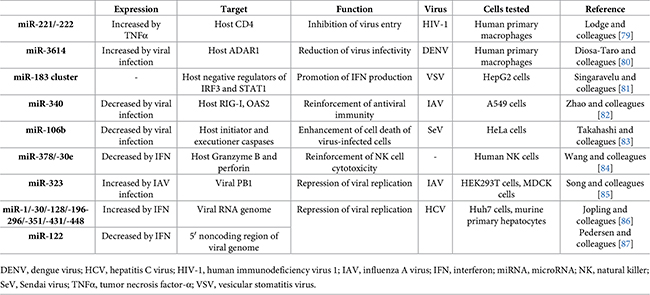
Inmunidad adaptativa tipo CRISPR dirigida por piRNA
Los piRNA son ARN pequeños que suelen tener entre 24 y 31nt, y la mayoría se transcriben a partir de regiones conocidas como grupos de piRNA. Cabe indicar que la biogénesis, la composición de nucleótidos y el patrón de modificación de muchos piRNA son distintos de los de otros ARN pequeños. Los piRNA maduros forman complejos de silenciamiento inducido por piRNA (piRISC) con proteínas Piwi, y estos complejos pueden inducir la formación de heterocromatina o escindir directamente su ARN diana. Como los objetivos de piRISC suelen ser transcripciones de elementos transponibles (TE) y los defectos en el sistema de piRNA suelen provocar fallos en la gametogénesis, se considera que la función canónica de los piRNA es el mantenimiento de la integridad del genoma de las células germinales. Hasta ahora, se han encontrado piRNA auténticos de mamíferos en las gónadas, aunque las proteínas Piwi y/o los ARN pequeños que comparten características de los piRNA se han asociado con funciones en algunos tejidos somáticos, como la regulación de la inmunidad innata en los pulmones.
Las fuentes de piRNA antivirales incluyen el procesamiento directo de ARN virales exógenos, fragmentos de ARN (p. ej., ARNt) o transcripciones de elementos virales endógenos (EVE), incluidos aquellos dentro de grupos de piRNA. Alrededor del 9% del genoma de un humano está representado por EVE, que son secuencias derivadas de virus que han sufrido transferencia horizontal de genes (HGT) en la línea germinal de nuestros ancestros. La producción de piRNA antivirales a partir de EVEs recuerda a una estrategia inmunitaria adaptativa en procariotas, el sistema (CRISPR)/asociado a CRISPR (Cas). En ambos casos, las secuencias de elementos móviles “no propios” se incorporan al genoma por HGT para funcionar en la memoria inmunitaria.
En muchas células somáticas o cancerosas, las manipulaciones epigenéticas que dan como resultado la expresión de ERV también inducen la señalización de IFN de una manera dependiente de RLR. En células germinales deficientes en piRNA, la regulación positiva de ERV y otras TE se asocia con el arresto meiótico y la apoptosis, aunque las señales precisas que desencadenan estos defectos siguen sin estar claras. Puede ser necesaria una vía de piRNA intacta para evitar que los ERV, expresados inevitablemente durante los borrados epigenéticos inherentes al desarrollo de las células germinales, desencadenen vías inmunitarias innatas incompatibles con la supervivencia de las células germinales. En particular, en las células progenitoras neuronales, la expresión de TE tras la desmetilación del ADN, se combina con una drástica regulación positiva de los genes de biogénesis de piRNA, lo que sugiere que algunas células somáticas pueden permanecer preparadas para generar piRNA tras una perturbación.
ARNt y fragmentos de ARNt (tRF) como moléculas antivirales
Los ARNt regulan las infecciones virales y los TE. Los virus generalmente no codifican ARNt, mientras que, en los humanos, los ARNt se encuentran entre los ARN más abundantes y también poseen una considerable diversidad. Los ARNt tienen una longitud de 70 a 90nt y pueden ser procesados en tRF (que por lo general varían de 14 a 32nt) por ribonucleasas, incluidas la angiogenina y Dicer, de una manera dependiente del estímulo. Los tRF se derivan de varios puntos a lo largo de la estructura en forma de hoja de trébol del ARNt y las mitades de tRF derivadas de los extremos 5′ y 3′ (tRF-5 y tRF-3, respectivamente) regulan la infección viral.
Los retrovirus endógenos y exógenos, así como los TE derivados de ellos, utilizan ARNt como cebadores para iniciar la transcripción inversa. Los tRF pueden influir indirectamente en la replicación de los no retrovirus; y aunque informes sugieren la participación de tRF específicos en infección por virus, los cambios globales en los ARNt y los tRF también están vinculados a la inmunidad antiviral. Recientemente, se ha demostrado que la protección de los ARNt de la escisión inducida por estrés por la angiogenina en las células T da como resultado una mayor susceptibilidad a la infección viral.
Se informa que algunos tRF se cargan en las proteínas Ago o Piwi. Además, se ha demostrado que varios guían el silenciamiento específico de secuencia de las transcripciones celulares y virales. Asimismo, las mitades de tRF sirven como una fuente importante de piRNA derivado de tRNA (td-piRNA) en el gusano de seda, pero la biogénesis de td-piRNA en mamíferos aún no se ha aclarado por completo.
Aplicaciones actuales y futuras de los ARN pequeños como medicamentos de ácidos nucleicos
Los ARN pequeños tienen características favorables como terapias antivirales. La FDA ha aprobado varios tipos de ARN de 18 a 30nt de longitud como medicamentos de ácidos nucleicos a partir de abril de 2021: 9 oligonucleótidos antisentido (ASO), 1 aptámero, 4 ARNi y 1 oligonucleótido CpG. Las secuencias y modificaciones específicas de estos medicamentos de ácidos nucleicos no están presentes en la naturaleza, pero cada uno aprovecha un sistema endógeno dirigido por ARN pequeños.
Como se demostró de manera convincente en el desarrollo de vacunas de ARNm contra COVID-19, que tomó solo 45 días desde la publicación de la secuencia preliminar del genoma del SARS-CoV-2 hasta el envío de un producto para ensayos clínicos, los medicamentos de ácidos nucleicos se pueden sintetizar químicamente en grandes cantidades rápidamente.
El reconocimiento objetivo y no objetivo de los ARN pequeños se puede diseñar racionalmente y analizar in silico, en comparación con bases de datos de ácidos nucleicos presentes en células humanas no infectadas. Esto representa una ventaja sobre las plataformas existentes para diseñar terapias basadas en proteínas, en particular los anticuerpos monoclonales, a pesar de los avances en la predicción de la estructura y las simulaciones de dinámica molecular.
La administración de medicamentos de ácido nucleico a sus tejidos objetivo fue un obstáculo inicial importante; los ARN pequeños se degradan rápidamente en el suero por las ARNasas o están sujetos a filtración glomerular. Sin embargo, los avances recientes en los sistemas de administración de fármacos, como las nanopartículas lipídicas o la glicosilación de ácidos nucleicos, han abordado estos desafíos, como lo ha demostrado nuevamente la vacunación con ARNm.
La capacidad de personalización de los ácidos nucleicos es otra característica prometedora para la medicina individualizada. Por ejemplo, se diseñó y utilizó un ASO personalizado, Milasen, para un paciente con enfermedad de Batten. Esto requirió menos de un año desde la secuenciación del genoma del paciente, lo que permitió un diseño racional, hasta la mejora de los síntomas.
De manera similar a la secuenciación del genoma humano, que está pasando a un uso clínico de rutina, la infraestructura de secuenciación de virus aumentará sustancialmente, por lo que el diseño de ARN pequeños basados en los genotipos virales que circulan en un individuo o población se está volviendo más realista.
Finalmente, del mismo modo a los que se están desarrollando para el cáncer, los ARN pequeños que se dirigen a mutaciones patógenas conocidas podrían usarse para anticipar y limitar la evolución viral.
Conclusión sobre los sistemas antivirales dirigidos por ARN pequeño en mamíferos
El descubrimiento de los mecanismos que permiten la inmunidad humana a los virus ha dado lugar a muchas historias de éxito en el desarrollo de terapias antivirales, desde compuestos derivados de plantas hasta terapias con IFN, antagonistas del CCR5 y anticuerpos monoclonales.
Es probable que la aclaración del papel de los sistemas antivirales dirigidos por ARN pequeño en los mamíferos proporcione éxitos adicionales; las acciones antivirales descritas recientemente de los miRNA, piRNA y tRNA son especialmente prometedoras.
En paralelo a los avances en la ciencia básica, las terapias y profilácticos basados en ARNm han experimentado un desarrollo acelerado desde la pandemia de SARS-CoV-2, superando aparentemente las barreras previas a los medicamentos con ácidos nucleicos. Los ARN pequeños tienen un gran potencial para las futuras aplicaciones terapéuticas.
Fuente de este artículo
Este artículo fue publicado originalmente en la revista PLOS Pathogens, bajo la referencia: Takahashi T, Heaton SM, Parrish NF (2021) Mammalian antiviral systems directed by small RNA. PLoS Pathog 17(12): e1010091. https://doi.org/10.1371/journal.ppat.1010091; estando disponible con acceso libre en la propia web de PLOS Pathogens.


